Al-Zahraa Mamdouh1† y Eman Zahran2*†
1Departamento de Enfermedades de los Peces, Instituto Nacional de Oceanografía y Pesca (NIOF), Egipto
2Departamento de Medicina de Animales Acuáticos, Facultad de Veterinaria, Universidad de Mansoura, Mansoura 35516, Egipto
†Estos autores comparten la autoría completa
*Autor correspondiente: [email protected] ORCID: 0000-0003-2212-3688
Los hongos presentes en alimentos o piensos producen micotoxinas que pueden dañar a las personas y los animales.

Los efectos hepatotóxicos, nefrotóxicos, carcinogénicos y mutagénicos de las micotoxinas, así como su impacto en los sistemas inmunitario y reproductor, las convierten en una de las principales preocupaciones en materia de seguridad de alimentos y piensos.
Los métodos tradicionales de detoxificación física y química de micotoxinas para alimentos y piensos pueden provocar la pérdida de nutrientes, la presencia de residuos de reactivos y la contaminación del medio ambiente. Así pues, el control de la contaminación por micotoxinas requiere nuevos métodos de detoxificación.
En este escenario, la tecnología de detoxificación microbiana se ha hecho cada vez más popular para degradar micotoxinas en alimentos y piensos debido a su alta eficiencia, baja toxicidad y fuerte especificidad sin deteriorar los nutrientes.
El azote de las micotoxinas en alimentos y piensos
Las micotoxinas son compuestos químicos tóxicos producidos como metabolitos secundarios por hongos filamentosos que contaminan los alimentos/piensos o las materias primas utilizadas para fabricarlos (Tola and Kebede, 2016).
Aspergillus, Penicillium y Fusarium representan los principales géneros de hongos micotoxigénicos, además de Trichoderma, Trichothecium y Alternaria (Ashiq, 2015).
El crecimiento de los mohos y la producción de micotoxinas vienen determinados por ciertas condiciones ambientales, entre las que se incluyen:
 Niveles de humedad (>70%)
Niveles de humedad (>70%) Temperatura (20–30 °C)
Temperatura (20–30 °C) Contenido de humedad de las plantas o cultivos (> 15%)
Contenido de humedad de las plantas o cultivos (> 15%) Daños mecánicos y por insectos en los cultivos y uso de plaguicidas
Daños mecánicos y por insectos en los cultivos y uso de plaguicidas Diversidad vegetal
Diversidad vegetal Carga de esporas fúngicas
Carga de esporas fúngicas
(Streit et al., 2012)

 La proliferación de diversos hongos en los productos agrícolas reduce el rendimiento y la calidad, con importantes pérdidas económicas (Adejumo and Adejoro, 2014).
La proliferación de diversos hongos en los productos agrícolas reduce el rendimiento y la calidad, con importantes pérdidas económicas (Adejumo and Adejoro, 2014).
Aproximadamente el 25% de los cultivos del mundo se contaminan anualmente con micotoxinas (Alshannaq and Yu, 2017).

Además, las pérdidas anuales causadas por la contaminación de materias primas con micotoxinas en las industrias ganadera (Ogunade et al., 2018) y acuícola (Anater et al., 2016) alcanzan mil millones de toneladas métricas de alimentos (Smith et al., 2016).
Anualmente, Estados Unidos se enfrenta a unas pérdidas económicas estimadas en 932 millones de dólares debido a las aflatoxinas, las fumonisinas y el deoxinivalenol (Bhatnagar et al., 2004).
La contaminación de alimentos y piensos con micotoxinas disminuye su valor nutricional y representa un riesgo potencial para la salud humana, animal y de los peces (Koletsi et al., 2021).
Biorremediación – Unaestrategia prometedorafrente a las micotoxinas
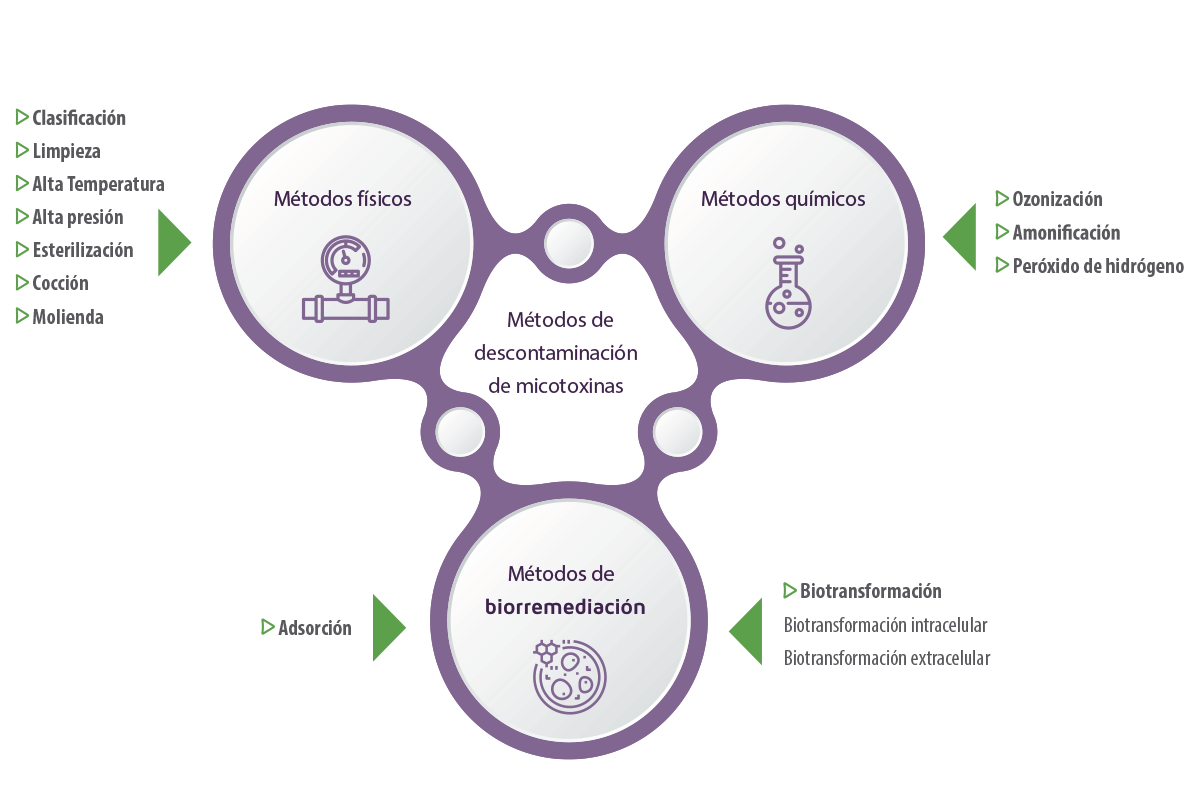
Durante mucho tiempo, los métodos de descontaminación de micotoxinas en piensos se han limitado a técnicas físicas y químicas.
- ⇰ Los métodos físicos incluyen la clasificación, la limpieza, la alta temperatura, la alta presión y otras prácticas, como la esterilización, la cocción y la molienda (Grenier et al., 2014; Pankaj et al., 2018).
- ⇰ Los métodos químicos incluyen el uso de agentes químicos para reducir o convertir las micotoxinas en subproductos menos tóxicos a través de la ozonización, la amoniación y el uso de peróxido de hidrógeno (Čolović et al., 2019).
Aunque estos métodos han reducido eficazmente la contaminación por micotoxinas, tienen limitaciones, como el hecho de no ser respetuosos con el medio ambiente, los elevados costes operativos y los resultados menos fiables. Además, pueden disminuir la calidad o el valor nutricional de los alimentos (Čolović et al., 2019; Pankaj et al., 2018).
 La biorremediación es una prometedora tecnología emergente para la degradación y detoxificación de micotoxinas basada en el uso de entidades biológicas, como bacterias u hongos, para detoxificar o eliminar toxinas ambientales (Chandra, 2015).
La biorremediación es una prometedora tecnología emergente para la degradación y detoxificación de micotoxinas basada en el uso de entidades biológicas, como bacterias u hongos, para detoxificar o eliminar toxinas ambientales (Chandra, 2015).Convertir las micotoxinas en compuestos no tóxicos utilizando agentes microbianos podría ser la mejor alternativa a los métodos clásicos de descontaminación.
 La biorremediación se considera uno de los enfoques más prometedores porque tiene una alta eficiencia de degradación y conlleva menos riesgos para el medio ambiente y una menor reducción de la calidad de los alimentos y piensos.
La biorremediación se considera uno de los enfoques más prometedores porque tiene una alta eficiencia de degradación y conlleva menos riesgos para el medio ambiente y una menor reducción de la calidad de los alimentos y piensos.
La descontaminación de los alimentos mediante enzimas microbianas es aún más favorable, ya que se ha producido un importante desarrollo de las tecnologías microbianas y de ingeniería genética y un rápido progreso en el estudio de la degradación de micotoxinas por enzimas clave y enzimas degradadoras expresadas de forma heterogénea.
Además, estas enzimas son específicas para cada sustrato, eficaces y respetuosas con el medio ambiente, y en general pueden mantener el valor alimentario sin causar pérdida de nutrientes ni de textura de los alimentos (Wang y Xie, 2020).
Mecanismos de biorremediación de micotoxinas
Los microorganismos pueden degradar las micotoxinas mediante adsorción o biotransformación.
- ⇰ La adsorción implica la unión física de las micotoxinas a polisacáridos y proteínas en las estructuras celulares externas del microrganismo, formando un complejo toxinamicrorganismo que puede excretarse (Nazareth et al., 2019; Sadiq et al., 2019).
- ⇰ La biotransformación implica la conversión de micotoxinas en compuestos no tóxicos o menos tóxicos bajo la acción de los microbios o de sus enzimas intracelulares y extracelulares (Alberts et al., 2009; Vega et al., 2017).
La biodegradación intracelular se basa en el tratamiento metabólico normal de las moléculas por los microorganismos dentro de la célula (Sandlin et al., 2022), mientras que la biotransformación extracelular se produce mediante la producción de enzimas estables que pueden degradar las micotoxinas.
Esta tecnología básica permite obtener varios productos comerciales (Moll, 2019).

Micotoxinas comunes y su biorremediación
AFLATOXINAS
Las aflatoxinas (AFB) son las micotoxinas más peligrosas, tóxicas y estudiadas.
Producidas principalmente por Aspergillus flavus y A. parasiticus y, en menor medida, por A. nomius, A. bombycis, A. pseudotamari y A. ochraceoroseus (Varga et al., 2011), se clasifican según su fluorescencia azul o verde bajo luz ultravioleta en AFB1, AFB2, AFG1 y AFG2 (Dhanasekaran et al., 2011).
Se producen en diversos alimentos para animales, como el maíz, los frutos secos molidos, las semillas de algodón de arroz y sorgo, las especias, los cereales, la soja, el cacao y la carne (Patriarca y Pinto, 2017; Vila-Donat et al., 2018).
Las aflatoxinas tienen propiedades hepatotóxicas, teratogénicas, mutagénicas (Kucukcakan y Hayrulai- Musliu, 2015; Rieswijk et al., 2016; Rotimi et al., 2017) e inmunosupresoras (Gao et al., 2023; Gong et al., 2016; Mohsenzadeh et al., 2016).
 La AFB1 es uno de los hepatocarcinógenos más potentes y uno de los principales responsables de la aparición de hepatocarcinomas (HCC) en todo el mundo (Kew, 2013).
La AFB1 es uno de los hepatocarcinógenos más potentes y uno de los principales responsables de la aparición de hepatocarcinomas (HCC) en todo el mundo (Kew, 2013).Biorremediación de aflatoxinas mediante adsorción microbiana
Existen numerosos estudios sobre la biodegradación de las aflatoxinas, ya sea por agentes microbianos o por sus enzimas.
Entre los microorganismos que pueden unirse a las aflatoxinas y eliminarlas por adsorción, los probióticos, como las bacterias ácido-lácticas y las levaduras, son los más estudiados porque pueden añadirse durante la fermentación de los alimentos para degradar las aflatoxinas en el proceso de producción (Shetty y Jespersen, 2006).
BACTERIAS ÁCIDO-LÁCTICAS
 Lactobacillus rhamnosus (GG) eliminó el 54% de AFB1 del medio de jugo intestinal de pollo en 1 min (El-Nezami et al., 2000).
Lactobacillus rhamnosus (GG) eliminó el 54% de AFB1 del medio de jugo intestinal de pollo en 1 min (El-Nezami et al., 2000). Las bacterias ácido-lácticas redujeron, in vitro, el transporte, el metabolismo y la toxicidad de la AFB1 en células Caco-2 (Gratz et al., 2007).
Las bacterias ácido-lácticas redujeron, in vitro, el transporte, el metabolismo y la toxicidad de la AFB1 en células Caco-2 (Gratz et al., 2007). En un estudio sobre la unión de 20 cepas de bacterias ácido-lácticas y bifidobacterias a la AFB1 en una solución contaminada se observó que dos cepas de Lactobacillus amylovorus y una cepa de L. rhamnosus se unieron a más del 50% de AFB1 a las 72 h (Peltonen et al., 2001).
En un estudio sobre la unión de 20 cepas de bacterias ácido-lácticas y bifidobacterias a la AFB1 en una solución contaminada se observó que dos cepas de Lactobacillus amylovorus y una cepa de L. rhamnosus se unieron a más del 50% de AFB1 a las 72 h (Peltonen et al., 2001). Lactobacillus plantarum C88 aislada de alimentos fermentados tradicionales chinos redujo la toxicidad de la AFB1 en ratones, lo que demuestra que el mecanismo detrás de la detoxificación fue una fuerte capacidad de unión que resultó en una mayor cantidad de AFB1 no absorbida en las heces (Huang et al., 2017).
Lactobacillus plantarum C88 aislada de alimentos fermentados tradicionales chinos redujo la toxicidad de la AFB1 en ratones, lo que demuestra que el mecanismo detrás de la detoxificación fue una fuerte capacidad de unión que resultó en una mayor cantidad de AFB1 no absorbida en las heces (Huang et al., 2017).
 Se descubrió que L. plantarum MON03 (LP) aislada de mantequilla artesanal tunecina mostraba una capacidad de unión significativa a AFB1 y AFM1 en solución salina amortiguadora de fosfato (PBS) en un 82% y 89%, respectivamente, en 24 h de incubación con la capacidad de tolerar la acidez gástrica y fuertes propiedades hidrofílicas de la superficie de las células, y eficacia de adherencia a las células Caco-3 in vitro (Jebali et al., 2015).
Se descubrió que L. plantarum MON03 (LP) aislada de mantequilla artesanal tunecina mostraba una capacidad de unión significativa a AFB1 y AFM1 en solución salina amortiguadora de fosfato (PBS) en un 82% y 89%, respectivamente, en 24 h de incubación con la capacidad de tolerar la acidez gástrica y fuertes propiedades hidrofílicas de la superficie de las células, y eficacia de adherencia a las células Caco-3 in vitro (Jebali et al., 2015). L. plantarum MON03 y L. rhamnosus GAF01 fueron aislados y probados por sus capacidades para eliminar AFM1 de PBS y leche reconstituida y se encontró que las dos cepas eliminaron AFM1, tanto en PBS y leche desnatada después de 0, 6, y 24 h a 37 °C, y sus capacidades de unión oscilaron entre 16,1-78,6% y 15,3-95,1%, respectivamente (Abbès et al., 2013).
L. plantarum MON03 y L. rhamnosus GAF01 fueron aislados y probados por sus capacidades para eliminar AFM1 de PBS y leche reconstituida y se encontró que las dos cepas eliminaron AFM1, tanto en PBS y leche desnatada después de 0, 6, y 24 h a 37 °C, y sus capacidades de unión oscilaron entre 16,1-78,6% y 15,3-95,1%, respectivamente (Abbès et al., 2013).
LEVADURAS
 Saccharomyces cerevisiae fijó el 90,3 ± 0,3% y el 92,7 ± 0,7% de AFM1 durante 30 y 60 min, respectivamente, en la leche. En comparación, fue capaz de fijar el 100% de AFM1 en 60 minutos cuando se combinó con un grupo de otras cepas de bacterias ácido-lácticas (L. rhamnosus, Lactobacillus delbrueckii spp. bulgaricus y Bifidobacterium lactis) (Corassin et al., 2013).
Saccharomyces cerevisiae fijó el 90,3 ± 0,3% y el 92,7 ± 0,7% de AFM1 durante 30 y 60 min, respectivamente, en la leche. En comparación, fue capaz de fijar el 100% de AFM1 en 60 minutos cuando se combinó con un grupo de otras cepas de bacterias ácido-lácticas (L. rhamnosus, Lactobacillus delbrueckii spp. bulgaricus y Bifidobacterium lactis) (Corassin et al., 2013). S. cerevisiae fijó el 60% y el 40% de AFB1 en residuos de fermentación de cerveza a pH 3 y 6,5, respectivamente (Campagnollo et al., 2015).
S. cerevisiae fijó el 60% y el 40% de AFB1 en residuos de fermentación de cerveza a pH 3 y 6,5, respectivamente (Campagnollo et al., 2015). Al evaluar la capacidad de los productos a base de S. cerevisiae procedentes de la fermentación de la caña de azúcar (levadura autolizada y levadura seca) para ligar AFB1 en PBS se vio que la levadura autolizada ligaba entre el 90,4% y el 97,5% de AFB1. La capacidad de fijación de la levadura seca fue del 96,5-99,3% (Gonçalves et al., 2015).
Al evaluar la capacidad de los productos a base de S. cerevisiae procedentes de la fermentación de la caña de azúcar (levadura autolizada y levadura seca) para ligar AFB1 en PBS se vio que la levadura autolizada ligaba entre el 90,4% y el 97,5% de AFB1. La capacidad de fijación de la levadura seca fue del 96,5-99,3% (Gonçalves et al., 2015).
 Chlebicz y Śliżewska (2020) estudiaron la capacidad de 12 cepas de Lactobacillus sp. y seis cepas de levadura S. cerevisiae para ligar AFB1, DON, fumonisina B1 y toxina T-2.
Chlebicz y Śliżewska (2020) estudiaron la capacidad de 12 cepas de Lactobacillus sp. y seis cepas de levadura S. cerevisiae para ligar AFB1, DON, fumonisina B1 y toxina T-2.-
Descubrieron que todas las cepas detoxificaban las micotoxinas, con la mayor reducción de la concentración para la mezcla de FB1 y FB2, que oscilaba entre el 62 y el 77% para las cepas bacterianas y entre el 67 y el 74% para la levadura, y que el DON era la micotoxina más resistente. Su concentración se redujo entre un 19 y un 39% por cepas de Lactobacillus sp y entre un 22 y un 43% por levaduras tras 24 h de incubación, mientras que las tasas de detoxificación para la AFB1 y la toxina T-2 fueron del 60% y el 61% por Lactobacillus, respectivamente, y del 65% y el 69% por levaduras, respectivamente.
 S. cerevisiae NRRL Y-1089 fue capaz de eliminar el 87,20% de AFB1 tras 30 minutos de incubación y la fijación mejoró al aumentar el tiempo de incubación (Hathout y Abdel-Nasser, 2022).
S. cerevisiae NRRL Y-1089 fue capaz de eliminar el 87,20% de AFB1 tras 30 minutos de incubación y la fijación mejoró al aumentar el tiempo de incubación (Hathout y Abdel-Nasser, 2022).
Biorremediación de aflatoxinas mediante degradación microbiana
BACTERIAS
 Myxococcus fulvus ANSM068 aislado de heces de ciervo transformó el 80,7% de AFB1 en medio líquido VY/2 tras incubación a 30 °C durante 72 h (Guan et al., 2010).
Myxococcus fulvus ANSM068 aislado de heces de ciervo transformó el 80,7% de AFB1 en medio líquido VY/2 tras incubación a 30 °C durante 72 h (Guan et al., 2010). Bacillus velezensis DY3108 degradó AFB1 en un 91,5% con una temperatura óptima de 80 °C y un pH amplio (4,0 a 11,0) con la mayor degradación (94,70%) a pH 8,0 (Shu et al., 2018).
Bacillus velezensis DY3108 degradó AFB1 en un 91,5% con una temperatura óptima de 80 °C y un pH amplio (4,0 a 11,0) con la mayor degradación (94,70%) a pH 8,0 (Shu et al., 2018). Bacillus licheniformis CFR1 degradó AFB1 en un 94,7% tras 24 h de incubación con una temperatura óptima de 37 °C y pH 7 (Rao et al., 2017).
Bacillus licheniformis CFR1 degradó AFB1 en un 94,7% tras 24 h de incubación con una temperatura óptima de 37 °C y pH 7 (Rao et al., 2017).
 Stenotrophomonas acidoaminiphila CW117 degradó el 76,7% de la AFB1 a altas temperaturas (alcanzó los 90 ⁰C) y a pH 6-7, y la degradación dependió de la dosis y del tiempo (Cai et al., 2020).
Stenotrophomonas acidoaminiphila CW117 degradó el 76,7% de la AFB1 a altas temperaturas (alcanzó los 90 ⁰C) y a pH 6-7, y la degradación dependió de la dosis y del tiempo (Cai et al., 2020). Una Escherichia coli CG1061 no patógena eliminó la AFB1 con una tasa de degradación del 93,7% a una temperatura óptima de 55 °C y un pH 8,5 (Wang et al., 2019a).
Una Escherichia coli CG1061 no patógena eliminó la AFB1 con una tasa de degradación del 93,7% a una temperatura óptima de 55 °C y un pH 8,5 (Wang et al., 2019a). Pseudomonas aeruginosa N17-1 degradó AFB1, AFB2 y AFM1 en un 82,8%, 46,8% y 31,9% después de la incubación en medio de caldo nutriente (NB) a 37 °C durante 72 h, con una degradación máxima a 55 °C y después de la adición de manganeso (Mn2+) y cobre (Cu2+) como activadores (Sangare et al., 2014).
Pseudomonas aeruginosa N17-1 degradó AFB1, AFB2 y AFM1 en un 82,8%, 46,8% y 31,9% después de la incubación en medio de caldo nutriente (NB) a 37 °C durante 72 h, con una degradación máxima a 55 °C y después de la adición de manganeso (Mn2+) y cobre (Cu2+) como activadores (Sangare et al., 2014).
HONGOS
 Aspergillus niger RAF106 detoxificó eficazmente AFB1 a compuestos con baja o ninguna mutagenicidad en ambientes con valores de pH de 4-10 y temperaturas de 25-45 °C (Fang et al., 2020).
Aspergillus niger RAF106 detoxificó eficazmente AFB1 a compuestos con baja o ninguna mutagenicidad en ambientes con valores de pH de 4-10 y temperaturas de 25-45 °C (Fang et al., 2020). A. niger FS10 degradó la AFB1 en un 98% a un compuesto menos tóxico tras 12 h de incubación (Fang et al., 2020).
A. niger FS10 degradó la AFB1 en un 98% a un compuesto menos tóxico tras 12 h de incubación (Fang et al., 2020).
Biorremediación de aflatoxinas mediante degradación enzimática
 Las aflatoxinas son degradadas por una enzima reductasa de Mycobacterium smegmatis que puede utilizar el cofactor deazaflavina F420H2 y catalizar la reducción de la fracción éster α,β-insaturada de las aflatoxinas, activando las moléculas para su hidrólisis espontánea y detoxificación (Taylor et al., 2010).
Las aflatoxinas son degradadas por una enzima reductasa de Mycobacterium smegmatis que puede utilizar el cofactor deazaflavina F420H2 y catalizar la reducción de la fracción éster α,β-insaturada de las aflatoxinas, activando las moléculas para su hidrólisis espontánea y detoxificación (Taylor et al., 2010). La manganeso peroxidasa producida por el hongo de la podredumbre blanca Phanerochaete sordida YK-624 fue capaz de detoxificar la AFB1 mediante una reacción de oxidación que consistía en primer lugar en la oxidación a AFB1-8,9-epóxido por MnP y después en la hidrólisis a AFB1– 8,9-dihidrodio (Wang et al., 2011).
La manganeso peroxidasa producida por el hongo de la podredumbre blanca Phanerochaete sordida YK-624 fue capaz de detoxificar la AFB1 mediante una reacción de oxidación que consistía en primer lugar en la oxidación a AFB1-8,9-epóxido por MnP y después en la hidrólisis a AFB1– 8,9-dihidrodio (Wang et al., 2011). El tratamiento del maíz contaminado con filtrados de cultivos de Trametes versicolor que contienen enzimas ligninolíticas redujo significativamente el contenido de AFB1 (Scarpari et al., 2014).
El tratamiento del maíz contaminado con filtrados de cultivos de Trametes versicolor que contienen enzimas ligninolíticas redujo significativamente el contenido de AFB1 (Scarpari et al., 2014). La manganeso peroxidasa, que puede iniciar la despolimerización oxidativa de la lignina en hongos degradadores de lignocelulosa Irpex lacteus, Phanerochaete chrysosporium, Ceriporiopsis subvermispora y Nematoloma forward, fue capaz de degradar AFB1, ZEN, DON y FB1 (Wang et al., 2019d).
La manganeso peroxidasa, que puede iniciar la despolimerización oxidativa de la lignina en hongos degradadores de lignocelulosa Irpex lacteus, Phanerochaete chrysosporium, Ceriporiopsis subvermispora y Nematoloma forward, fue capaz de degradar AFB1, ZEN, DON y FB1 (Wang et al., 2019d). La capacidad de la lacasa C30 de S. cerevisiae para degradar cuatro aflatoxinas (AFB1, AFB2, AFG1 y AFG2), mostrando que la capacidad de unión de las aflatoxinas a la enzima era AFB1>AFG2>AFG1>AFB2.
La capacidad de la lacasa C30 de S. cerevisiae para degradar cuatro aflatoxinas (AFB1, AFB2, AFG1 y AFG2), mostrando que la capacidad de unión de las aflatoxinas a la enzima era AFB1>AFG2>AFG1>AFB2.-
También se demostró que las aflatoxinas pueden interactuar cerca del centro de cobre T1 de la enzima a través de enlaces H e interacciones hidrofóbicas con residuos de aminoácidos. Además, la lacasa fue capaz de degradar todas las aflatoxinas, y la actividad de degradación aumentó a medida que el tiempo de incubación aumentó de 12 a 60 h, con una degradación máxima para AFB1, AFB2, AFG1 y AFG2 de 90,33%, 74,23%, 85,24%, y 87,58%, respectivamente, a 30 °C en 0,1 M PBS y pH 5,7 (Liu et al., 2020).
 Una enzima lacasa del hongo Pleurotus pulmonarius degradó AFB1 en condiciones óptimas de pH 7 y una temperatura de 37 °C (Song et al., 2021).
Una enzima lacasa del hongo Pleurotus pulmonarius degradó AFB1 en condiciones óptimas de pH 7 y una temperatura de 37 °C (Song et al., 2021). Una lacasa de Bacillus amyloliquefaciens B10 fue separada y se encontró que degradaba 79,3% de AFB1 en 36 h bajo una temperatura óptima de 40 °C y pH 6-8 con la adición de magnesio (Mg2+) y dimetil sulfóxido (DMSO) como potenciadores de la reacción (Xiong et al., 2022).
Una lacasa de Bacillus amyloliquefaciens B10 fue separada y se encontró que degradaba 79,3% de AFB1 en 36 h bajo una temperatura óptima de 40 °C y pH 6-8 con la adición de magnesio (Mg2+) y dimetil sulfóxido (DMSO) como potenciadores de la reacción (Xiong et al., 2022). Se clonó un gen de lacasa a partir de Bacillus vallismortis fmb-103 y se expresó en el hospedador heterólogo E. coli tras la optimización del codón y se investigó su degradación de AFB1, encontrándose que mejoraba con el aumento del pH y la temperatura de incubación y superaba el 60% a pH 7 y 37 °C (Bian et al., 2022).
Se clonó un gen de lacasa a partir de Bacillus vallismortis fmb-103 y se expresó en el hospedador heterólogo E. coli tras la optimización del codón y se investigó su degradación de AFB1, encontrándose que mejoraba con el aumento del pH y la temperatura de incubación y superaba el 60% a pH 7 y 37 °C (Bian et al., 2022). Una enzima degradadora de AFB1 producida por Pseudomonas aeruginosa degradó AFB1 en condiciones óptimas de 65 °C y pH 6 con la presencia de Cu2+ y hierro (Fe3+) (Song et al., 2019).
Una enzima degradadora de AFB1 producida por Pseudomonas aeruginosa degradó AFB1 en condiciones óptimas de 65 °C y pH 6 con la presencia de Cu2+ y hierro (Fe3+) (Song et al., 2019). Las proteínas o enzimas degradadoras de AFB1 en los extractos intracelulares de A. niger RAF106 indujeron una degradación del 80% de AFB1 durante 24 h de incubación (Fang et al., 2020).
Las proteínas o enzimas degradadoras de AFB1 en los extractos intracelulares de A. niger RAF106 indujeron una degradación del 80% de AFB1 durante 24 h de incubación (Fang et al., 2020). La aflatoxina-oxidasa producida por Armillariella tabescens, un hongo comestible y medicinal chino, fue eficaz en la degradación de AFB1 actuando sobre el anillo bisfurano de AFB1 que se considera la estructura tóxica clave (Cao et al., 2011).
La aflatoxina-oxidasa producida por Armillariella tabescens, un hongo comestible y medicinal chino, fue eficaz en la degradación de AFB1 actuando sobre el anillo bisfurano de AFB1 que se considera la estructura tóxica clave (Cao et al., 2011).
OCRATOXINAS
Las ocratoxinas (OT) son micotoxinas extremadamente tóxicas.
La ocratoxina A (OTA), el tipo más frecuente encontrado en alimentos y piensos es producida principalmente por Aspergillus ochraceus, mientras que la ocratoxina B y C son producidas por otras especies de Aspergillus y Penicillium (Marroquín-Cardona et al., 2014).
La OTA es la más tóxica y prevalente de la familia de las ocratoxinas (Heussner y Bingle, 2015).
Es nefrotóxica (Khoi et al., 2021), genotóxica, citotóxica, teratogénica (Hibi et al., 2011), carcinogénica (Pfohl-Leszkowicz y Manderville, 2012), mutagénica, inmunotóxica y mielotóxica (Marin y Taranu, 2015).
Biorremediación de la OTA mediante adsorción microbiana
Se ha descrito la capacidad de diversos microorganismos para eliminar la OTA mediante adsorción. La capacidad de los microorganismos para adsorber OTA se ve afectada por muchos factores, entre ellos:
 Cepas microbianas (su estado es viable o inviable (Bejaoui et al., 2004; Piotrowska, 2014).
Cepas microbianas (su estado es viable o inviable (Bejaoui et al., 2004; Piotrowska, 2014). Condiciones de cultivo, incluidos el pH y la temperatura (Gil-Serna et al., 2011).
Condiciones de cultivo, incluidos el pH y la temperatura (Gil-Serna et al., 2011).
BACTERIAS ÁCIDO-LÁCTICAS
 En un estudio reciente, se investigó la detoxificación de la OTA utilizando cultivos individuales y conjuntos de Lactobacillus acidophilus y L. rhamnosus en diversos modos, incluidas células viables, calentadas e inactivadas por ultrasonidos, así como una combinación de estas condiciones, y los resultados fueron diferentes en función de la estructura de la pared celular, la hidrofobicidad y la capacidad de unión de toxinas de las bacterias lácticas individuales y conjuntas (Abedi et al., 2022).
En un estudio reciente, se investigó la detoxificación de la OTA utilizando cultivos individuales y conjuntos de Lactobacillus acidophilus y L. rhamnosus en diversos modos, incluidas células viables, calentadas e inactivadas por ultrasonidos, así como una combinación de estas condiciones, y los resultados fueron diferentes en función de la estructura de la pared celular, la hidrofobicidad y la capacidad de unión de toxinas de las bacterias lácticas individuales y conjuntas (Abedi et al., 2022).
 Se investigó la capacidad de unión de tres especies de bacterias lácticas térmicamente activas o inactivadas, Lactobacillus brevis, L. plantarum y Lactobacillus sanfranciscensis, y se señaló que la unión de las células bacterianas inactivadas era mayor que la de las células viables, y la adsorción de OTA a la pared celular se atribuyó a interacciones hidrofóbicas y electrostáticas (Piotrowska, 2014).
Se investigó la capacidad de unión de tres especies de bacterias lácticas térmicamente activas o inactivadas, Lactobacillus brevis, L. plantarum y Lactobacillus sanfranciscensis, y se señaló que la unión de las células bacterianas inactivadas era mayor que la de las células viables, y la adsorción de OTA a la pared celular se atribuyó a interacciones hidrofóbicas y electrostáticas (Piotrowska, 2014). Se aislaron 54 bacterias ácido-lácticas de muestras de trigo almacenadas y se estudió su capacidad para eliminar la OTA en un medio líquido, y los resultados dependieron de la temperatura, el pH y la biomasa bacteriana en cultivos mixtos, con un alto porcentaje de reducción de OTA obtenido por L. plantarum y Lactobacillus graminis (más del 97%), seguidos de Pediococcus pentosaceus (más del 81,5%) (Belkacem-Hanfi et al., 2014).
Se aislaron 54 bacterias ácido-lácticas de muestras de trigo almacenadas y se estudió su capacidad para eliminar la OTA en un medio líquido, y los resultados dependieron de la temperatura, el pH y la biomasa bacteriana en cultivos mixtos, con un alto porcentaje de reducción de OTA obtenido por L. plantarum y Lactobacillus graminis (más del 97%), seguidos de Pediococcus pentosaceus (más del 81,5%) (Belkacem-Hanfi et al., 2014).
 P. pentosaceus (RG7B) eliminó el 84% de la OTA en medios MRS, mientras que su capacidad de unión en medios PBS fue sólo del 25% (Taroub et al., 2019).
P. pentosaceus (RG7B) eliminó el 84% de la OTA en medios MRS, mientras que su capacidad de unión en medios PBS fue sólo del 25% (Taroub et al., 2019). Se probaron catorce cepas aisladas de BAL y Bifidobacterium para evaluar su capacidad para unirse a la OTA en PB contaminadas con 10 ppb de OTA. Estas cepas probadas redujeron entre un 80,4 y un 81,4% la OTA a 37 ⁰C en 36 h (Fouad et al., 2021).
Se probaron catorce cepas aisladas de BAL y Bifidobacterium para evaluar su capacidad para unirse a la OTA en PB contaminadas con 10 ppb de OTA. Estas cepas probadas redujeron entre un 80,4 y un 81,4% la OTA a 37 ⁰C en 36 h (Fouad et al., 2021).
LEVADURAS
 Bejaoui et al. (2004) señalaron que las levaduras tratadas con calor y ácido podían adsorber el 90,8% y el 73% de la OTA, respectivamente, mientras que las células viables fijaban hasta el 35% de la OTA.
Bejaoui et al. (2004) señalaron que las levaduras tratadas con calor y ácido podían adsorber el 90,8% y el 73% de la OTA, respectivamente, mientras que las células viables fijaban hasta el 35% de la OTA.-
En el mismo estudio, la capacidad de adsorción bajo tratamiento térmico o ácido se atribuyó a su capacidad para alterar las características superficiales de las células microbianas, como la ampliación del tamaño de los poros, la desnaturalización de las proteínas y la formación de productos de reacción de Maillard entre polisacáridos y péptidos.
 Se evaluaron las capacidades de eliminación de OTA de cuatro cepas de S. cerevisiae y los resultados mostraron que las eficiencias de adsorción de OTA variaban según las cepas de levadura y las concentraciones de OTA. Además, hubo una correlación entre la capacidad de eliminación de micotoxinas y la cantidad de pared celular, lo que sugiere que la interacción levadura-micotoxina es más de tipo adsorción (Armando et al., 2012).
Se evaluaron las capacidades de eliminación de OTA de cuatro cepas de S. cerevisiae y los resultados mostraron que las eficiencias de adsorción de OTA variaban según las cepas de levadura y las concentraciones de OTA. Además, hubo una correlación entre la capacidad de eliminación de micotoxinas y la cantidad de pared celular, lo que sugiere que la interacción levadura-micotoxina es más de tipo adsorción (Armando et al., 2012).
 S. cerevisiae (ambas cepas Syrena LOCK 0201 y Málaga LOCK 0173) disminuyeron la cantidad de OTA.
S. cerevisiae (ambas cepas Syrena LOCK 0201 y Málaga LOCK 0173) disminuyeron la cantidad de OTA.-
La cepa Málaga LOCK 0173 fue capaz de eliminar el 82,8% y el 10,7% de los medios de uva y grosella negra, respectivamente, mientras que la capacidad de fijación de la cepa Syrena LOCK 0201 fue del 85,1% para uva y del 65,2% para grosella negra (Piotrowska et al., 2013).
 Dos cepas de S. cerevisiae disminuyeron el nivel de OTA un 70% en 3 días a 30°C en presencia de 250 g l-1 de azúcares y con fosfato diamónico (Petruzzi et al., 2014).
Dos cepas de S. cerevisiae disminuyeron el nivel de OTA un 70% en 3 días a 30°C en presencia de 250 g l-1 de azúcares y con fosfato diamónico (Petruzzi et al., 2014).

OTROS MICROORGANISMOS
 Se reportó que una cepa de Bacillus megaterium JSW-B1 fijó 62.6% de OTA en medio de cultivo líquido dentro de 72 h y el tratamiento térmico aumentó notablemente la capacidad de fijación de OTA de las células, indicando que la actividad de eliminación de OTA de esta cepa resultó principalmente de la adsorción a las células bacterianas (Shang et al., 2019).
Se reportó que una cepa de Bacillus megaterium JSW-B1 fijó 62.6% de OTA en medio de cultivo líquido dentro de 72 h y el tratamiento térmico aumentó notablemente la capacidad de fijación de OTA de las células, indicando que la actividad de eliminación de OTA de esta cepa resultó principalmente de la adsorción a las células bacterianas (Shang et al., 2019).
 El uso de Bacillus subtilis inmovilizado en perlas de alginato de sodio comestible (pH 3 y 25% de cloruro de sodio, NaCl) fue altamente eficaz en la reducción de OTA en el vino tinto, donde redujo el 90% de la OTA del cultivo y cuando estas perlas se añadieron al vino tinto enriquecido con OTA, redujo la OTA en un 78,50 ± 0,49% después de 1 h (Shukla et al., 2020).
El uso de Bacillus subtilis inmovilizado en perlas de alginato de sodio comestible (pH 3 y 25% de cloruro de sodio, NaCl) fue altamente eficaz en la reducción de OTA en el vino tinto, donde redujo el 90% de la OTA del cultivo y cuando estas perlas se añadieron al vino tinto enriquecido con OTA, redujo la OTA en un 78,50 ± 0,49% después de 1 h (Shukla et al., 2020).
![]() Recientemente, se ha demostrado que la adsorción de OTA a microorganismos se controla genéticamente. Por ejemplo:
Recientemente, se ha demostrado que la adsorción de OTA a microorganismos se controla genéticamente. Por ejemplo:
 L. delbrueckii NRRL B-1024 se mutagenizó utilizando metanosulfonato de etilo (EMS) para inducir variaciones genéticas con el fin de mejorar la biosorción de OTA. Siete de 19 mutantes mostraron una biosorción de OTA superior a la cepa de tipo salvaje (Khattab et al., 2018).
L. delbrueckii NRRL B-1024 se mutagenizó utilizando metanosulfonato de etilo (EMS) para inducir variaciones genéticas con el fin de mejorar la biosorción de OTA. Siete de 19 mutantes mostraron una biosorción de OTA superior a la cepa de tipo salvaje (Khattab et al., 2018). Se utilizaron 12 cepas de S. cerevisiae para obtener híbridos intraespecíficos y cinco de los 27 híbridos validados mostraron una mayor actividad de adsorción de OTA, pero menor de compuestos fenólicos en comparación con sus progenitores (Caridi et al., 2020).
Se utilizaron 12 cepas de S. cerevisiae para obtener híbridos intraespecíficos y cinco de los 27 híbridos validados mostraron una mayor actividad de adsorción de OTA, pero menor de compuestos fenólicos en comparación con sus progenitores (Caridi et al., 2020).
Biorremediación de la OTA mediante degradación microbiana
La degradación microbiana de la OTA se produce a través de dos reacciones catalizadas por enzimas:
 Hidrólisis del enlace amida entre el residuo de isocumarina y la fenilalanina.
Hidrólisis del enlace amida entre el residuo de isocumarina y la fenilalanina. Hidrólisis del anillo de lactona.
Hidrólisis del anillo de lactona.
Entre los productos de degradación notificados, la OTα se considera no tóxica o menos tóxica (Bittner et al., 2015; Haq et al., 2016) y varias cepas bacterianas y fúngicas han demostrado su capacidad para degradar la OTA con o sin producción de OTα.
BACTERIAS
 B. licheniformis Sl-1 degradó la OTA en medios líquidos en los que las células esterilizadas en autoclave fijaron un 80% de OTA mientras que las células viables un 60% de OTA tras 24 h de incubación aeróbica.
B. licheniformis Sl-1 degradó la OTA en medios líquidos en los que las células esterilizadas en autoclave fijaron un 80% de OTA mientras que las células viables un 60% de OTA tras 24 h de incubación aeróbica.-
El sobrenadante libre de células de Sl-1 degradó la OTA con una tasa de degradación del 98% tras 24 h de incubación aeróbica. No se produjeron productos de degradación y, cuando se aplicó a muestras de maíz enmohecido, la tasa de degradación de la OTA fue del 35% (Shi et al., 2013).
 La OTA fue degradada por células viables y autoclavadas de B. subtilis CW 14 en más del 60% y fue degradada por el sobrenadante libre de células de CW 14 en un 97,6% de tasa de degradación después de 24 h de incubación a 30 °C sin productos de degradación producidos. Además, mediante la inoculación del cultivo se degradó el 47,1% de la OTA en muestras de maíz contaminadas (Shi et al., 2014).
La OTA fue degradada por células viables y autoclavadas de B. subtilis CW 14 en más del 60% y fue degradada por el sobrenadante libre de células de CW 14 en un 97,6% de tasa de degradación después de 24 h de incubación a 30 °C sin productos de degradación producidos. Además, mediante la inoculación del cultivo se degradó el 47,1% de la OTA en muestras de maíz contaminadas (Shi et al., 2014). B. velezensis E2 degradó el 96,1% de la OTA en 48 h por transformación enzimática e hidrólisis alcalina produciendo ocratoxina α abierta (OP-OTα) como producto de degradación (Zhang et al., 2022).
B. velezensis E2 degradó el 96,1% de la OTA en 48 h por transformación enzimática e hidrólisis alcalina produciendo ocratoxina α abierta (OP-OTα) como producto de degradación (Zhang et al., 2022).
HONGOS
 Las cepas Aspergillus tubingensis M036 y M074 degradaron la OTA en más de un 95% después de 14 días, produciendo ocratoxina α.
Las cepas Aspergillus tubingensis M036 y M074 degradaron la OTA en más de un 95% después de 14 días, produciendo ocratoxina α.-
Además, las enzimas crudas de los cultivos de ambas cepas condujeron a la biodegradación de OTA en un 97,5% y 91,3% a pH 5 y 80,3% y 75,3% a pH 7, respectivamente, después de 24 h (Cho et al., 2016).
 A niger W-35 degradó la OTA in vitro y en piensos comerciales tras incubación a 37 °C durante 12 h en un 78% y 37%, respectivamente, con una producción de OTα.
A niger W-35 degradó la OTA in vitro y en piensos comerciales tras incubación a 37 °C durante 12 h en un 78% y 37%, respectivamente, con una producción de OTα.-
Además, una enzima especí ica para la degradación de la OTA (ocratoxinasa, OTasa) obtenida de W-35 se expresó con éxito en E. coli BL21 y degradó la OTA a una tasa del 85,1% durante 12 h (Zhao et al., 2020).
 Una nueva cepa de Aspergillus oryzae fue capaz de degradar el 94% de la OTA tras 72 h de incubación (Xiong et al., 2021). Además, la cepa 396.1 de Aspergillus calcoaceticus y la cepa neg1 de Acinetobacter sp. fueron capaces de convertir el 82% y el 91% de OTA en OTα en 6 días de incubación a 24 °C (De Bellis et al., 2015).
Una nueva cepa de Aspergillus oryzae fue capaz de degradar el 94% de la OTA tras 72 h de incubación (Xiong et al., 2021). Además, la cepa 396.1 de Aspergillus calcoaceticus y la cepa neg1 de Acinetobacter sp. fueron capaces de convertir el 82% y el 91% de OTA en OTα en 6 días de incubación a 24 °C (De Bellis et al., 2015). Lactobacillus johnsonii CECT 289, L. rhamnosus CECT 278T y L. plantarum CECT 749 presentaron valores de degradación del 97,4 %, 97 % y 95 % de la OTA en OTα y fenilalanina (Phe) (Luz et al., 2018).
Lactobacillus johnsonii CECT 289, L. rhamnosus CECT 278T y L. plantarum CECT 749 presentaron valores de degradación del 97,4 %, 97 % y 95 % de la OTA en OTα y fenilalanina (Phe) (Luz et al., 2018).
Biorremediación de la OTA mediante degradación enzimática
 La carboxipeptidasa, una enzima hidrolizada relacionada con la conversión enzimática de la OTA, se clonó a partir del genoma de B. amyloliquefaciens ASAG1 utilizando el sistema de expresión de E. coli y se informó de que tenía una elevada actividad degradadora de la OTA (Chang et al., 2015).
La carboxipeptidasa, una enzima hidrolizada relacionada con la conversión enzimática de la OTA, se clonó a partir del genoma de B. amyloliquefaciens ASAG1 utilizando el sistema de expresión de E. coli y se informó de que tenía una elevada actividad degradadora de la OTA (Chang et al., 2015). El gen de la carboxipeptidasa dacA de B. subtilis CW14 expresado en Escherichia coli, contribuyó a degradar la OTA en un 71,3% (Xu et al., 2021).
El gen de la carboxipeptidasa dacA de B. subtilis CW14 expresado en Escherichia coli, contribuyó a degradar la OTA en un 71,3% (Xu et al., 2021). La OTA se degradó bajo la actividad de la carboxipeptidasa PJ_1540 producida por Acinetobacter sp. neg1, ITEM 17016 (Liuzzi et al., 2017).
La OTA se degradó bajo la actividad de la carboxipeptidasa PJ_1540 producida por Acinetobacter sp. neg1, ITEM 17016 (Liuzzi et al., 2017).
 Otro estudio investigó la capacidad de la enzima lacasa producida por Pleurotus eryngii y los sistemas lacasamediador (LMS) para degradar la OTA, y se encontró que degradaba el 27% de OTA (Loi et al., 2018).
Otro estudio investigó la capacidad de la enzima lacasa producida por Pleurotus eryngii y los sistemas lacasamediador (LMS) para degradar la OTA, y se encontró que degradaba el 27% de OTA (Loi et al., 2018). Otra enzima de la que se ha informado que es capaz de degradar la OTA es la ocratoxinasa de A. niger.
Otra enzima de la que se ha informado que es capaz de degradar la OTA es la ocratoxinasa de A. niger.-
En un estudio realizado por Dobritzsch et al. (2014) se clonó un gen putativo de amidasa que codifica un polipéptido de 480 aminoácidos y se expresó de forma homóloga en A. niger. Se cree que la proteína recombinante es más eficiente en la hidrólisis de la ocratoxina A que la enzima carboxipeptidasa.
ZEARALENONA
La zearalenona (ZEN) es una micotoxina estrogénica no esteroidea producida principalmente por Fusarium graminearum y, en menor medida, por Fusarium culmorum, Fusarium cerealis o Fusarium equiseti (De Boevre et al., 2012; Taheur et al., 2017).
La ZEN se produce de forma natural en el maíz, la cebada, la avena, el trigo, el arroz, el sorgo, el centeno y otros productos básicos importantes cuando las condiciones de humedad y temperatura son adecuadas (Golge y Kabak, 2020).
Puede activar los receptores estrogénicos, lo que provoca trastornos reproductivos en animales (Gupta, 2012) y especies de peces (Schwartz et al., 2013; Woźny et al., 2020).
La ZEN y algunos de sus derivados también pueden inducir efectos genotóxicos mediante la inducción de aductos de ADN, fragmentación del ADN, apoptosis, micronúcleos y aberraciones cromosómicas (Gao et al., 2013).
Biorremediación del ZEN mediante adsorción microbiana
La ZEN puede adsorberse a las paredes celulares microbianas a través de sus proteínas, lípidos y carbohidratos (manosa, peptidoglicanos, glucanos).
Los mecanismos de adsorción incluyen interacciones iónicas, enlaces de hidrógeno e interacciones hidrofóbicas (Wang et al., 2019b) y dependen de muchos factores, entre ellos:
 Cepas microbianas: la presencia de carbohidratos en su pared celular y su grosor.
Cepas microbianas: la presencia de carbohidratos en su pared celular y su grosor. Condiciones de cultivo: pH y temperatura, tiempo de incubación y adición de potenciadores físicos y químicos.
Condiciones de cultivo: pH y temperatura, tiempo de incubación y adición de potenciadores físicos y químicos.

SACCHAROMYCES
 La adición de 0,2% de adsorbente de pared celular de levadura al maíz contaminado con ZEN alivió eficazmente la toxicidad reproductiva de los lechones (Shang et al., 2016).
La adición de 0,2% de adsorbente de pared celular de levadura al maíz contaminado con ZEN alivió eficazmente la toxicidad reproductiva de los lechones (Shang et al., 2016). 10 cepas de S. cerevisiae y 5 de Saccharomyces pastorianus pudieron adsorber 30-70% de ZEN dependiendo de la cepa en mosto contaminado después de un periodo de fermentación de 96 h (Wall-Martínez et al., 2019).
10 cepas de S. cerevisiae y 5 de Saccharomyces pastorianus pudieron adsorber 30-70% de ZEN dependiendo de la cepa en mosto contaminado después de un periodo de fermentación de 96 h (Wall-Martínez et al., 2019). Se investigó la capacidad del residuo de fermentación de cerveza que contenía S. cerevisiae para fijar el ZEN a diferentes niveles de pH y se observó que el 75,1% y el 77,5% del ZEN se eliminaban a pH 3 y 6,5, respectivamente (Campagnollo et al., 2015).
Se investigó la capacidad del residuo de fermentación de cerveza que contenía S. cerevisiae para fijar el ZEN a diferentes niveles de pH y se observó que el 75,1% y el 77,5% del ZEN se eliminaban a pH 3 y 6,5, respectivamente (Campagnollo et al., 2015).
 La capacidad de S. cerevisiae para disminuir el contenido de ZEN puede verse influida por el pretratamiento físico o químico.
La capacidad de S. cerevisiae para disminuir el contenido de ZEN puede verse influida por el pretratamiento físico o químico.-
Por ejemplo, la capacidad máxima de adsorción de S. cerevisiae incubada en condiciones libres de estrés fue del 36,5%. En comparación, la incubación en condiciones de estrés (inducida por nanopartículas de plata y liofilización) aumentó el valor máximo de sorción al 57,0% (Rogowska et al., 2019).
 La relación diámetro celular/espesor de la pared celular mostró una correlación entre la cantidad de pared celular y la capacidad de eliminación de ZEN, y hubo un aumento significativo en la unión a ZEN después de la exposición a condiciones gastrointestinales (Armando et al., 2012).
La relación diámetro celular/espesor de la pared celular mostró una correlación entre la cantidad de pared celular y la capacidad de eliminación de ZEN, y hubo un aumento significativo en la unión a ZEN después de la exposición a condiciones gastrointestinales (Armando et al., 2012).-
Se ha sugerido que S. cerevisiae puede unir micotoxinas a su pared celular a través de los carbohidratos funcionales que contiene (polímeros de glucomanano).
 Se ha informado de que la pared celular de S. cerevisiae podría unirse a ZEN y se encontró que el β(1,3)-d-glucano y el β(1,6)-d-glucano eran las estructuras más favorables para la adsorción de zearalenona (Yiannikouris et al., 2004).
Se ha informado de que la pared celular de S. cerevisiae podría unirse a ZEN y se encontró que el β(1,3)-d-glucano y el β(1,6)-d-glucano eran las estructuras más favorables para la adsorción de zearalenona (Yiannikouris et al., 2004). En un estudio más reciente, S. cerevisiae NRRL Y-1089 y S. cerevisiae NRRL Y-1089 eliminaron ZEN en un 94,80% y 91,80%, respectivamente, y la eliminación de ZEN disminuyó al aumentar el período de incubación (Hathout y Abdel-Nasser, 2022).
En un estudio más reciente, S. cerevisiae NRRL Y-1089 y S. cerevisiae NRRL Y-1089 eliminaron ZEN en un 94,80% y 91,80%, respectivamente, y la eliminación de ZEN disminuyó al aumentar el período de incubación (Hathout y Abdel-Nasser, 2022).
BACTERIAS ÁCIDO-LÁCTICAS
 Se evaluó la capacidad de ocho cepas de Lactobacillus pentosus para adsorber ZEN de una solución tampón de acetato sódico. La capacidad de adsorción fue del 83,17%, 82,78% y 81,69% para las cepas JM0812, UM054 y UM055, respectivamente (Sangsila et al., 2016).
Se evaluó la capacidad de ocho cepas de Lactobacillus pentosus para adsorber ZEN de una solución tampón de acetato sódico. La capacidad de adsorción fue del 83,17%, 82,78% y 81,69% para las cepas JM0812, UM054 y UM055, respectivamente (Sangsila et al., 2016). Las cepas GG y LC705 de L. rhamnosus podían unir el 55% de ZEN y α-ZOL, y se informó de que ambas toxinas podrían compartir el mismo sitio de unión en la superficie bacteriana (El-Nezami et al., 2002).
Las cepas GG y LC705 de L. rhamnosus podían unir el 55% de ZEN y α-ZOL, y se informó de que ambas toxinas podrían compartir el mismo sitio de unión en la superficie bacteriana (El-Nezami et al., 2002). En un estudio más reciente, Lactococcus lactis y Bifidobacterium sp fueron capaces de adsorber ZEN en un 90 % y 88 %, respectivamente, mediante la unión a grupos carboxilo desprotonados (principalmente de asparagina y glutamina) de las proteínas y el peptidoglicano de BAL (Król et al., 2018).
En un estudio más reciente, Lactococcus lactis y Bifidobacterium sp fueron capaces de adsorber ZEN en un 90 % y 88 %, respectivamente, mediante la unión a grupos carboxilo desprotonados (principalmente de asparagina y glutamina) de las proteínas y el peptidoglicano de BAL (Król et al., 2018).
 L. rhamnosus y otras especies podrían absorber del 40% al 68,2% de ZEN, y estas cepas podrían formar un enlace permanente con ZEN, lo que inspiró el diseño de un nuevo polvo liofilizado basado en manchas de LAB para la desintoxicación de ZEN (Vega et al., 2017).
L. rhamnosus y otras especies podrían absorber del 40% al 68,2% de ZEN, y estas cepas podrían formar un enlace permanente con ZEN, lo que inspiró el diseño de un nuevo polvo liofilizado basado en manchas de LAB para la desintoxicación de ZEN (Vega et al., 2017). La adsorción de ZEN por L. lactis no fue homogénea, donde se expresó con una etapa inicial rápida con alrededor del 90% de biosorción de ZEN y con un segundo paso mucho más lento, mientras que la adsorción por Bifidobacterium sp fue homogénea en un 88% (Król et al., 2017).
La adsorción de ZEN por L. lactis no fue homogénea, donde se expresó con una etapa inicial rápida con alrededor del 90% de biosorción de ZEN y con un segundo paso mucho más lento, mientras que la adsorción por Bifidobacterium sp fue homogénea en un 88% (Król et al., 2017). Se observó que otras cepas de BAL adsorbían múltiples micotoxinas. Se informó de que Lactobacillus kefiri, Kazachstania servazzii y Acetobacter syzygii adsorbían ZEN, AFB1 y OTA, donde L. kefiri KFLM3 fue la más activa, adsorbiendo del 80 al 100% de las micotoxinas cuando se cultivó en leche, mientras que la cepa K. servazzii KFGY7 adsorbía el 65, 69 y 67% para AFB1, OTA y ZEN, respectivamente (Taheur et al., 2017).
Se observó que otras cepas de BAL adsorbían múltiples micotoxinas. Se informó de que Lactobacillus kefiri, Kazachstania servazzii y Acetobacter syzygii adsorbían ZEN, AFB1 y OTA, donde L. kefiri KFLM3 fue la más activa, adsorbiendo del 80 al 100% de las micotoxinas cuando se cultivó en leche, mientras que la cepa K. servazzii KFGY7 adsorbía el 65, 69 y 67% para AFB1, OTA y ZEN, respectivamente (Taheur et al., 2017). En la misma línea, las especies Streptococcus y Enterococcus fueron capaces de ligar hasta el 33, 49, 24 y 62% de DON, ZEN, FB1 y FB2, respectivamente (Niderkorn et al., 2007).
En la misma línea, las especies Streptococcus y Enterococcus fueron capaces de ligar hasta el 33, 49, 24 y 62% de DON, ZEN, FB1 y FB2, respectivamente (Niderkorn et al., 2007).
Biorremediación del ZEN mediante degradación microbiana
 ZEN fue completamente degradada por S. cerevisiae tras 48 h de incubación y se sugirió que los mecanismos implicados en la degradación eran la producción de enzimas intracelulares y extracelulares y algunas proteínas funcionales producidas por S. cerevisiae (Zhang et al., 2016).
ZEN fue completamente degradada por S. cerevisiae tras 48 h de incubación y se sugirió que los mecanismos implicados en la degradación eran la producción de enzimas intracelulares y extracelulares y algunas proteínas funcionales producidas por S. cerevisiae (Zhang et al., 2016). Varias cepas de S. cerevisiae fueron capaces de degradar más del 90% de ZEN del medio de cultivo en dos días con la producción de α- y β-zearalenol menos tóxicos (α-ZOL 53% y β-ZOL 8%) como productos finales, lo que sugiere que la eliminación se debió principalmente a la biotransformación del ZEN más que a su adsorción a las paredes de las células de levadura (Keller et al., 2015).
Varias cepas de S. cerevisiae fueron capaces de degradar más del 90% de ZEN del medio de cultivo en dos días con la producción de α- y β-zearalenol menos tóxicos (α-ZOL 53% y β-ZOL 8%) como productos finales, lo que sugiere que la eliminación se debió principalmente a la biotransformación del ZEN más que a su adsorción a las paredes de las células de levadura (Keller et al., 2015).
 B. subtilis ANSB01G degradó 84,58, 66,34 y 83,04% de ZEN en maíz naturalmente contaminado, granos secos de destilería con solubles y alimento completo para cerdos, respectivamente.
B. subtilis ANSB01G degradó 84,58, 66,34 y 83,04% de ZEN en maíz naturalmente contaminado, granos secos de destilería con solubles y alimento completo para cerdos, respectivamente.-
Se cree que la degradación fue enzimática porque los tratamientos con calentamiento o la adición de proteinasa K redujeron significativamente la tasa de degradación del ZEN en el sobrenadante del cultivo (Lei et al., 2014).
 B. amyloliquefaciens ZDS-1 degradó ZEN en un medio de cultivo y trigo mediante degradación enzimática sin producción de derivados de ZEN (Xu et al., 2016).
B. amyloliquefaciens ZDS-1 degradó ZEN en un medio de cultivo y trigo mediante degradación enzimática sin producción de derivados de ZEN (Xu et al., 2016). B. amyloliquefaciens LN disminuyó la concentración de ZEN en un 92% después de 36 h de incubación y se informó que B. amyloliquefaciens LN no solo poseía capacidad de adsorción de ZEN, sino que también exhibía la capacidad de degradar ZEN. Además, B. amyloliquefaciens LN no era hemolítica ni producía enterotoxinas (Lee et al., 2017).
B. amyloliquefaciens LN disminuyó la concentración de ZEN en un 92% después de 36 h de incubación y se informó que B. amyloliquefaciens LN no solo poseía capacidad de adsorción de ZEN, sino que también exhibía la capacidad de degradar ZEN. Además, B. amyloliquefaciens LN no era hemolítica ni producía enterotoxinas (Lee et al., 2017).
 Numerosas cepas de Bacillus fueron capaces de degradar ZEN, incluyendo B. licheniformis que disminuyó más del 98% de ZEN después de 36 h de incubación a través de mecanismos no hemolíticos, no productores de enterotoxinas mediante la producción de altos niveles de xilanasa extracelular, celulasa y actividades de proteasa (Yi et al., 2011).
Numerosas cepas de Bacillus fueron capaces de degradar ZEN, incluyendo B. licheniformis que disminuyó más del 98% de ZEN después de 36 h de incubación a través de mecanismos no hemolíticos, no productores de enterotoxinas mediante la producción de altos niveles de xilanasa extracelular, celulasa y actividades de proteasa (Yi et al., 2011). Las tasas de degradación de ZEN de B. subtilis y B. natto fueron del 100% y 87% en 48 h, respectivamente; los dos taxones tienen una alta actividad de degradación de ZEN en cerveza, harina de maíz y harina de soja (Ju et al., 2019).
Las tasas de degradación de ZEN de B. subtilis y B. natto fueron del 100% y 87% en 48 h, respectivamente; los dos taxones tienen una alta actividad de degradación de ZEN en cerveza, harina de maíz y harina de soja (Ju et al., 2019).
Biodegradación del ZEN por enzimas microbianas
Tres tipos principales de enzimas biológicas pueden degradar el ZEN: la lacasa, la lactona hidrolasa y la peroxidasa.
Atacan tres sitios diferentes de la molécula de ZEN:
- 1. La primera posición es el enlace éster del anillo de lactona, y si el anillo de lactona puede abrirse, la molécula resultante pierde sus propiedades de imitación del estrógeno.
- 2. El segundo sitio de reacción está en el largo anillo de lactona, especialmente en el enlace C-C cerca del grupo carbonilo.
- 3. La tercera implica la apertura del anillo aromático como forma de degradar el ZEN.
(Kriszt et al., 2012; Wu et al., 2021)
LACTONA HIDROLASAS
Las lactona hidrolasas son las más estudiadas para la degradación del ZEN.
 La lactona hidrolasa ZHD101 se extrajo del hongo micoparásito Clonostachys rosea y degradó el ZEN en productos menos tóxicos (Takahashi-Ando et al., 2002).
La lactona hidrolasa ZHD101 se extrajo del hongo micoparásito Clonostachys rosea y degradó el ZEN en productos menos tóxicos (Takahashi-Ando et al., 2002). Se descubrió que Gliocladium roseum produce lactona hidrolasa que puede romper e hidrolizar el enlace de lactona de ZEN para formar derivados de ZEN no tóxicos (Utermark y Karlovsky, 2007).
Se descubrió que Gliocladium roseum produce lactona hidrolasa que puede romper e hidrolizar el enlace de lactona de ZEN para formar derivados de ZEN no tóxicos (Utermark y Karlovsky, 2007). Bi et al. (2018) señalaron que la ZEN lactonasa de Neurospora crassa está codificada por el gen zenc y sobreexpresada en Pichia pastoris.
Bi et al. (2018) señalaron que la ZEN lactonasa de Neurospora crassa está codificada por el gen zenc y sobreexpresada en Pichia pastoris. Posteriormente, la enzima zenc purificada se añadió a los granos secos de destilería con solubles (DDGS), subproductos de maíz y salvado de maíz, y la concentración de ZEN disminuyó en un 71%, 89% y 95%, respectivamente.
Posteriormente, la enzima zenc purificada se añadió a los granos secos de destilería con solubles (DDGS), subproductos de maíz y salvado de maíz, y la concentración de ZEN disminuyó en un 71%, 89% y 95%, respectivamente.
LACASA
 La enzima lacasa se aisló de la especie fúngica T. versicolor y se observó que disminuía el contenido de ZEN en un medio líquido en un 81,7 % tras 4 h de incubación a su temperatura óptima (Banu et al., 2013).
La enzima lacasa se aisló de la especie fúngica T. versicolor y se observó que disminuía el contenido de ZEN en un medio líquido en un 81,7 % tras 4 h de incubación a su temperatura óptima (Banu et al., 2013). La lacasa CotA de B. subtilis (BsCotA) combinada con el compuesto químico metilsiringato degradó el ZEN en un 100 % (Wang et al., 2019c).
La lacasa CotA de B. subtilis (BsCotA) combinada con el compuesto químico metilsiringato degradó el ZEN en un 100 % (Wang et al., 2019c). Se investigó la capacidad de la lacasa (LC) de P. eryngii y los sistemas lacasa-mediador (LMS) para degradar el ZEN, y se encontró que el ZEN se degradaba en un 100% (Loi et al., 2018).
Se investigó la capacidad de la lacasa (LC) de P. eryngii y los sistemas lacasa-mediador (LMS) para degradar el ZEN, y se encontró que el ZEN se degradaba en un 100% (Loi et al., 2018). Una enzima lacasa del hongo Pleurotus pulmonarius fue capaz de degradar ZEN en condiciones óptimas de pH (4-8) y temperatura (40-60 ◦C) (Song et al., 2021).
Una enzima lacasa del hongo Pleurotus pulmonarius fue capaz de degradar ZEN en condiciones óptimas de pH (4-8) y temperatura (40-60 ◦C) (Song et al., 2021).
PEROXIDASA
Otros estudios han demostrado la capacidad de la enzima peroxidasa para degradar ZEN.
-
 Los extractos extracelulares del género Acinetobacter degradaron completamente la cepa SM04 de zearalenona en medio M1 tras 12 h de incubación (Yu et al., 2011b).
Los extractos extracelulares del género Acinetobacter degradaron completamente la cepa SM04 de zearalenona en medio M1 tras 12 h de incubación (Yu et al., 2011b).  Se extrajeron enzimas de Acinetobacter sp y se degradó la ZEN a productos no estrogénicos más pequeños.
Se extrajeron enzimas de Acinetobacter sp y se degradó la ZEN a productos no estrogénicos más pequeños.-
La enzima se identificó como peroxiredoxina, y el peróxido de hidrógeno oxidó el ZEN y sus productos mediante la catálisis de la peroxiredoxina (Yu et al., 2011a).
 Wang et al. (2019d) informaron de que ocho peroxidasas de manganeso de diferentes hongos degradadores de lignocelulosa podían degradar el 90,2-94,3% de ZEN en presencia de malonato, por lo que los radicales libres desempeñan un papel importante.
Wang et al. (2019d) informaron de que ocho peroxidasas de manganeso de diferentes hongos degradadores de lignocelulosa podían degradar el 90,2-94,3% de ZEN en presencia de malonato, por lo que los radicales libres desempeñan un papel importante.

Conclusiones
Las bacterias, las levaduras y los hongos pueden detoxificar las micotoxinas de forma eficaz y específica.
La degradación microbiana descompone las toxinas sin dejar residuos tóxicos para la seguridad de alimentos y piensos. Además, la degradación microbiana y la adsorción pueden convertir varias micotoxinas comunes en sustancias relativamente seguras, haciendo que los alimentos y los piensos sean más seguros.
Los estudios recientes sobre micotoxinas microbianas en alimentos o piensos se han centrado en la eliminación de micotoxinas individuales y en la aplicación de cepas en el laboratorio.
Esta revisión propone varias direcciones futuras:
- 1. En primer lugar, cepas detoxificadas o enzimas degradadoras de alimentos.
2. En segundo lugar, para mejorar la degradación y reducir la estabilidad de las micotoxinas, estudiar la combinación de múltiples cepas y la degradación microbiana de las micotoxinas.
REFERENCIAS
Abbès, S., Salah-Abbès, J.B., Sharafi, H., Jebali, R., Noghabi, K.A., Oueslati, R., 2013. Ability of Lactobacillus rhamnosus GAF01 to remove AFM1 in vitro and to counteract AFM1 immunotoxicity in vivo. Journal of Immunotoxicology 10, 279-286.
Abedi, E., Mousavifard, M., Hashemi, S.M.B., 2022. Ultrasound-Assisted Detoxification of Ochratoxin A: Comparative Study of Cell Wall Structure, Hydrophobicity, and Toxin Binding Capacity of Single and Co-culture Lactic Acid Bacteria. Food and Bioprocess Technology 15, 539-560.
Adejumo, T., Adejoro, D., 2014. Incidence of aflatoxins, fumonisins, trichothecenes and ochratoxins in Nigerian foods and possible intervention strategies. Food Science and Quality Management 31, 127-147.
Alberts, J., Gelderblom, W., Botha, A., Van Zyl, W., 2009. Degradation of aflatoxin B1 by fungal laccase enzymes. International journal of food microbiology 135, 47-52.
Alshannaq, A., Yu, J.-H., 2017. Occurrence, toxicity, and analysis of major mycotoxins in food. International journal of environmental research and public health 14, 632.
Anater, A., Manyes, L., Meca, G., Ferrer, E., Luciano, F.B., Pimpao, C.T., Font, G., 2016. Mycotoxins and their consequences in aquaculture: A review. Aquaculture 451, 1-10.
Armando, M., Pizzolitto, R., Dogi, C., Cristofolini, A., Merkis, C., Poloni, V., Dalcero, A., Cavaglieri, L.R., 2012. Adsorption of ochratoxin A and zearalenone by potential probiotic Saccharomyces cerevisiae strains and its relation with cell wall thickness. Journal of applied microbiology 113, 256-264.
Ashiq, S., 2015. Natural occurrence of mycotoxins in food and feed: Pakistan perspective. Comprehensive reviews in food science and food safety 14, 159-175.
Banu, I., Lupu, A., Aprodu, I., 2013. Degradation of zearalenone by laccase enzyme. Scientific Study & Research. Chemistry & Chemical Engineering, Biotechnology, Food Industry 14, 79.
Bejaoui, H., Mathieu, F., Taillandier, P., Lebrihi, A., 2004. Ochratoxin A removal in synthetic and natural grape juices by selected oenological Saccharomyces strains. Journal of applied microbiology 97, 1038-1044.
Belkacem-Hanfi, N., Fhoula, I., Semmar, N., Guesmi, A., Perraud-Gaime, I., Ouzari, H.-I., Boudabous, A., Roussos, S., 2014. Lactic acid bacteria against post-harvest moulds and ochratoxin A isolated from stored wheat. Biological Control 76, 52-59.
Bhatnagar, D., Payne, G.A., Cleveland, T.E., Robens, J.F. 2004. Mycotoxins: current issues in USA. In: Meeting the mycotoxin menace, 17-47.
Bi, K., Zhang, W., Xiao, Z., Zhang, D., 2018. Characterization, expression and application of a zearalenone degrading enzyme from Neurospora crassa. Amb Express 8, 1-10.
Bian, L., Zheng, M., Chang, T., Zhou, J., Zhang, C., 2022. Degradation of Aflatoxin B1 by recombinant laccase extracellular produced from Escherichia coli. Ecotoxicology and Environmental Safety 244, 114062.
Bittner, A., Cramer, B., Harrer, H., Humpf, H.-U., 2015. Structure elucidation and in vitro cytotoxicity of ochratoxin α amide, a new degradation product of ochratoxin A. Mycotoxin Research 31, 83-90.
Bueno, D.J., Casale, C.H., Pizzolitto, R.P., Salvano, M.A., Oliver, G., 2007. Physical adsorption of aflatoxin B1 by lactic acid bacteria and Saccharomyces cerevisiae: a theoretical model. Journal of food protection 70, 2148-2154.
Cai, M., Qian, Y., Chen, N., Ling, T., Wang, J., Jiang, H., Wang, X., Qi, K., Zhou, Y., 2020. Detoxification of aflatoxin B1 by Stenotrophomonas sp. CW117 and characterization the thermophilic degradation process. Environmental Pollution 261, 114178.
Campagnollo, F.B., Franco, L.T., Rottinghaus, G.E., Kobashigawa, E., Ledoux, D.R., Daković, A., Oliveira, C.A., 2015. In vitro evaluation of the ability of beer fermentation residue containing Saccharomyces cerevisiae to bind mycotoxins. Food research international 77, 643-648.
Cao, H., Liu, D., Mo, X., Xie, C., Yao, D., 2011. A fungal enzyme with the ability of aflatoxin B1 conversion: Purification and ESI-MS/MS identification. Microbiological Research 166, 475-483.
Caridi, A., Sidari, R., Pulvirenti, A., Blaiotta, G., 2020. Genetic improvement of wine yeasts for opposite adsorption activity of phenolics and ochratoxin A during red winemaking. Food Biotechnology 34, 352-370.
Chandra, R., 2015. Advances in biodegradation and bioremediation of industrial waste.
Chang, X., Wu, Z., Wu, S., Dai, Y., Sun, C., 2015. Degradation of ochratoxin A by Bacillus amyloliquefaciens ASAG1. Food Additives & Contaminants: Part A 32, 564-571.
Chlebicz, A., Śliżewska, K., 2020. In Vitro Detoxification of Aflatoxin B1, Deoxynivalenol, Fumonisins, T-2 Toxin and Zearalenone by Probiotic Bacteria from Genus Lactobacillus and Saccharomyces cerevisiae Yeast. Probiotics and Antimicrobial Proteins 12, 289-301.
Cho, S.M., Jeong, S.E., Lee, K.R., Sudhani, H.P., Kim, M., Hong, S.-Y., Chung, S.H., 2016. Biodegradation of ochratoxin A by Aspergillus tubingensis isolated from meju. Journal of Microbiology and Biotechnology 26, 1687-1695.
Čolović, R., Puvača, N., Cheli, F., Avantaggiato, G., Greco, D., Đuragić, O., Kos, J., Pinotti, L., 2019. Decontamination of mycotoxin-contaminated feedstuffs and compound feed. Toxins 11, 617.
Corassin, C.H., Bovo, F., Rosim, R.E., Oliveira, C.A.F.d., 2013. Efficiency of Saccharomyces cerevisiae and lactic acid bacteria strains to bind aflatoxin M1 in UHT skim milk. Food control 31, 80-83.
De Bellis, P., Tristezza, M., Haidukowski, M., Fanelli, F., Sisto, A., Mulè, G., Grieco, F., 2015. Biodegradation of ochratoxin A by bacterial strains isolated from vineyard soils. Toxins 7, 5079-5093.
De Boevre, M., Di Mavungu, J.D., Landschoot, S., Audenaert, K., Eeckhout, M., Maene, P., Haesaert, G., De Saeger, S., 2012. Natural occurrence of mycotoxins and their masked forms in food and feed products. World Mycotoxin Journal 5, 207-219.
Dhanasekaran, D., Shanmugapriya, S., Thajuddin, N., Panneerselvam, A., 2011. Aflatoxins and aflatoxicosis in human and animals. Aflatoxins- Biochemistry and Molecular Biology 10, 221-254.
Dobritzsch, D., Wang, H., Schneider, G., Yu, S., 2014. Structural and functional characterization of ochratoxinase, a novel mycotoxin-degrading enzyme. Biochemical Journal 462, 441-452.
El-Nezami, H., Mykkänen, H., Kankaanpää, P., Salminen, S., Ahokas, J., 2000. Ability of Lactobacillus and Propionibacterium strains to remove aflatoxin B1 from the chicken duodenum. Journal of food protection 63, 549-552.
El-Nezami, H., Polychronaki, N., Salminen, S., Mykkänen, H., 2002. Binding rather than metabolism may explain the interaction of two food-grade Lactobacillus strains with Zearalenone and Its Derivative ɑ́-Zearalenol. Applied and environmental microbiology 68, 3545-3549.
Fang, Q.a., Du, M., Chen, J., Liu, T., Zheng, Y., Liao, Z., Zhong, Q., Wang, L., Fang, X., Wang, J., 2020. Degradation and detoxification of aflatoxin B1 by tea-derived Aspergillus niger RAF106. Toxins 12, 777.
Fouad, M.T., El-Shenawy, M., El-Desouky, T.A., 2021. Efficiency of Selected Lactic Acid Bacteria Isolated from some Dairy Products on Aflatoxin [B.sub.1] and Ochratoxin A. Journal of Pure and Applied Microbiology 15, 312+.
Gao, F., Jiang, L.-p., Chen, M., Geng, C.-y., Yang, G., Ji, F., Zhong, L.-f., Liu, X.-f., 2013. Genotoxic effects induced by zearalenone in a human embryonic kidney cell line. Mutation Research/Genetic Toxicology and Environmental Mutagenesis 755, 6-10.
Gao, Y.-N., Wang, Z.-W., Yang, X., Wang, J.-Q., Zheng, N., 2023. Aflatoxin M1 and ochratoxin A induce a competitive endogenous RNA regulatory network of intestinal immunosuppression by whole transcriptome analysis. Science of The Total Environment 854, 158777.
Gil-Serna, J., Patiño, B., Cortés, L., González-Jaén, M.T., Vázquez, C., 2011. Mechanisms involved in reduction of ochratoxin A produced by Aspergillus westerdijkiae using Debaryomyces hansenii CYC 1244. International journal of food microbiology 151, 113-118.
Golge, O., Kabak, B., 2020. Occurrence of deoxynivalenol and zearalenone in cereals and cereal products from Turkey. Food Control 110, 106982.
Gonçalves, B.L., Rosim, R.E., de Oliveira, C.A.F., Corassin, C.H., 2015. The in vitro ability of different Saccharomyces cerevisiae–based products to bind aflatoxin B1. Food control 47, 298-300.
Gong, Y.Y., Watson, S., Routledge, M.N., 2016. Aflatoxin exposure and associated human health effects, a review of epidemiological studies. Food safety 4, 14-27.
Gratz, S., Wu, Q., El-Nezami, H., Juvonen, R., Mykkänen, H., Turner, P., 2007. Lactobacillus rhamnosus strain GG reduces aflatoxin B1 transport, metabolism, and toxicity in Caco-2 cells. Applied and environmental microbiology 73, 3958-3964.
Grenier, B., Loureiro‐Bracarense, A.P., Leslie, J.F., Oswald, I.P., 2014. Physical and chemical methods for mycotoxin decontamination in maize. Mycotoxin reduction in grain chains, 116-129.
Guan, S., Zhao, L., Ma, Q., Zhou, T., Wang, N., Hu, X., Ji, C., 2010. In vitro efficacy of Myxococcus fulvus ANSM068 to biotransform aflatoxin B1. International Journal of Molecular Sciences 11, 4063-4079.
Gupta, R.C., 2012. Veterinary toxicology: basic and clinical principles. Academic press. Haq, M., Gonzalez, N., Mintz, K., Jaja-Chimedza, A., De Jesus, C.L., Lydon, C., Welch, A.Z., Berry, J.P., 2016. Teratogenicity of ochratoxin A and the degradation product, ochratoxin α, in the zebrafish (Danio rerio) embryo model of vertebrate development. Toxins 8, 40.
Haskard, C.A., El-Nezami, H.S., Kankaanpää, P.E., Salminen, S., Ahokas, J.T., 2001. Surface binding of aflatoxin B1 by lactic acid bacteria. Applied and environmental microbiology 67, 3086-3091.
Hathout, A., Abdel-Nasser, A., 2022. The Efficiency of Saccharomyces Cerevisiae as an Antifungal and Antimycotoxigenic Agent.
Heussner, A.H., Bingle, L.E., 2015. Comparative ochratoxin toxicity: A review of the available data. Toxins 7, 4253-4282.
Hibi, D., Suzuki, Y., Ishii, Y., Jin, M., Watanabe, M., Sugita-Konishi, Y., Yanai, T., Nohmi, T., Nishikawa, A., Umemura, T., 2011. Site-Specific In V ivo Mutagenicity in the Kidney of gpt Delta Rats Given a Carcinogenic Dose of Ochratoxin A. Toxicological Sciences 122, 406-414.
Huang, L., Duan, C., Zhao, Y., Gao, L., Niu, C., Xu, J., Li, S., 2017. Reduction of aflatoxin B1 toxicity by Lactobacillus plantarum C88: a potential probiotic strain isolated from Chinese traditional fermented food “tofu”. PloS one 12, e0170109.
Jebali, R., Abbès, S., Salah-Abbès, J.B., Younes, R.B., Haous, Z., Oueslati, R., 2015. Ability of Lactobacillus plantarum MON03 to mitigate aflatoxins (B1 and M1) immunotoxicities in mice. Journal of immunotoxicology 12, 290-299.
Ju, J., Tinyiro, S.E., Yao, W., Yu, H., Guo, Y., Qian, H., Xie, Y., 2019. The ability of Bacillus subtilis and Bacillus natto to degrade zearalenone and its application in food. Journal of Food Processing and Preservation 43, e14122.
Keller, L., Abrunhosa, L., Keller, K., Rosa, C.A., Cavaglieri, L., Venâncio, A., 2015. Zearalenone and Its Derivatives α-Zearalenol and β-Zearalenol Decontamination by Saccharomyces cerevisiae Strains Isolated from Bovine Forage. Toxins 7, 3297-3308.
Kew, M.C., 2013. Aflatoxins as a cause of hepatocellular carcinoma. Journal of Gastrointestinal & Liver Diseases 22.
Khattab, A., Ibrahim, M., El-Kady, A., 2018. Ochratoxin A biosorption onto genetically improved of Lactobacillus delbrueckii mutants. International Food Research Journal 25.
Khoi, C.-S., Chen, J.-H., Lin, T.-Y., Chiang, C.-K., Hung, K.-Y., 2021. Ochratoxin A-induced nephrotoxicity: Up-to-date evidence. International Journal of Molecular Sciences 22, 11237.
Koletsi, P., Schrama, J.W., Graat, E.A., Wiegertjes, G.F., Lyons, P., Pietsch, C., 2021. The Occurrence of Mycotoxins in Raw Materials and Fish Feeds in Europe and the Potential Effects of Deoxynivalenol (DON) on the Health and Growth of Farmed Fish Species—A Review. Toxins 13, 403.
Kriszt, R., Krifaton, C., Szoboszlay, S., Cserháti, M., Kriszt, B., Kukolya, J., Czéh, Á., Fehér-Tóth, S., Török, L., Szőke, Z., 2012. A new zearalenone biodegradation strategy using non-pathogenic Rhodococcus pyridinivorans K408 strain.
Król, A., Pomastowski, P., Rafińska, K., Railean-Plugaru, V., Walczak, J., Buszewski, B., 2017. Microbiology neutralization of zearalenone using Lactococcus.
Król, A., Pomastowski, P., Rafińska, K., Railean-Plugaru, V., Walczak, J., Buszewski, B., 2018. Microbiology neutralization of zearalenone using Lactococcus lactis and Bifidobacterium sp. Analytical and bioanalytical chemistry 410, 943-952.
Kucukcakan, B., Hayrulai-Musliu, Z., 2015. Challenging role of dietary aflatoxin B1 exposure and hepatitis B infection on risk of hepatocellular carcinoma. Open access Macedonian journal of medical sciences 3, 363-369.
Lee, A., Cheng, K.-C., Liu, J.-R., 2017. Isolation and characterization of a Bacillus amyloliquefaciens strain with zearalenone removal ability and its probiotic potential. PloS one 12, e0182220.
Lei, Y., Zhao, L., Ma, Q., Zhang, J., Zhou, T., Gao, C., Ji, C., 2014. Degradation of zearalenone in swine feed and feed ingredients by Bacillus subtilis ANSB01G. World Mycotoxin Journal 7, 143-151.
Liu, Y., Mao, H., Hu, C., Tron, T., Lin, J., Wang, J., Sun, B., 2020. Molecular docking studies and in vitro degradation of four aflatoxins (AFB1, AFB2, AFG1, and AFG2) by a recombinant laccase from Saccharomyces cerevisiae. Journal of food science 85, 1353-1360.
Liuzzi, V.C., Fanelli, F., Tristezza, M., Haidukowski, M., Picardi, E., Manzari, C., Lionetti, C., Grieco, F., Logrieco, A.F., Thon, M.R., 2017. Transcriptional analysis of Acinetobacter sp. neg1 capable of degrading ochratoxin A. Frontiers in microbiology 7, 2162.
Loi, M., Fanelli, F., Cimmarusti, M.T., Mirabelli, V., Haidukowski, M., Logrieco, A.F., Caliandro, R., Mule, G., 2018. In vitro single and combined mycotoxins degradation by Ery4 laccase from Pleurotus eryngii and redox mediators. Food Control 90, 401-406.
Luz, C., Ferrer, J., Mañes, J., Meca, G., 2018. Toxicity reduction of ochratoxin A by lactic acid bacteria. Food and Chemical Toxicology 112, 60-66. Marin, D.E., Taranu, I., 2015. Ochratoxin A and its effects on immunity. Toxin Reviews 34, 11-20.
Marroquín-Cardona, A., Johnson, N., Phillips, T., Hayes, A., 2014. Mycotoxins in a changing global environment–a review. Food and Chemical Toxicology 69, 220-230.
Mohsenzadeh, M.S., Hedayati, N., Riahi-Zanjani, B., Karimi, G., 2016. Immunosuppression following dietary aflatoxin B1 exposure: a review of the existing evidence. Toxin Reviews 35, 121-127.
Moll, D., 2019. Enzyme technology for detoxification of mycotoxins in animal feed. Industrial Enzyme Applications, 219-254.
Nazareth, T.d.M., Luz, C., Torrijos, R., Quiles, J.M., Luciano, F.B., Mañes, J., Meca, G., 2019. Potential application of lactic acid bacteria to reduce aflatoxin B1 and fumonisin B1 occurrence on corn kernels and corn ears. Toxins 12, 21.
Niderkorn, V., Morgavi, D.P., Pujos, E., Tissandier, A., Boudra, H., 2007. Screening of fermentative bacteria for their ability to bind and biotransform deoxynivalenol, zearalenone and fumonisins in an in vitro simulated corn silage model. Food Additives & Contaminants 24, 406-415.
Ogunade, I., Martinez-Tuppia, C., Queiroz, O., Jiang, Y., Drouin, P., Wu, F., Vyas, D., Adesogan, A., 2018. Silage review: Mycotoxins in silage: Occurrence, effects, prevention, and mitigation. Journal of dairy science 101, 4034-4059.
Pankaj, S., Shi, H., Keener, K.M., 2018. A review of novel physical and chemical decontamination technologies for aflatoxin in food. Trends in Food Science & Technology 71, 73-83.
Patriarca, A., Pinto, V.F., 2017. Prevalence of mycotoxins in foods and decontamination. Current Opinion in Food Science 14, 50-60.
Peltonen, K., El-Nezami, H., Haskard, C., Ahokas, J., Salminen, S., 2001. Aflatoxin B1 binding by dairy strains of lactic acid bacteria and bifidobacteria. Journal of dairy science 84, 2152-2156.
Petruzzi, L., Bevilacqua, A., Baiano, A., Beneduce, L., Corbo, M.R., Sinigaglia, M., 2014. In vitro removal of ochratoxin A by two strains of Saccharomyces cerevisiae and their performances under fermentative and stressing conditions. Journal of Applied Microbiology 116, 60-70.
Pfohl-Leszkowicz, A., Manderville, R.A., 2012. An update on direct genotoxicity as a molecular mechanism of ochratoxin a carcinogenicity. Chemical research in toxicology 25, 252-262.
Piotrowska, M., 2014. The adsorption of ochratoxin A by Lactobacillus species. Toxins 6, 2826-2839.
Piotrowska, M., Nowak, A., Czyzowska, A., 2013. Removal of ochratoxin A by wine Saccharomyces cerevisiae strains. European Food Research and Technology 236, 441-447.
Raju, M., Devegowda, G., 2000. Influence of esterified-glucomannan on performance and organ morphology, serum biochemistry and haematology in broilers exposed to individual and combined mycotoxicosis (aflatoxin, ochratoxin and T-2 toxin). British poultry science 41, 640-650.
Rao, K.R., Vipin, A., Hariprasad, P., Appaiah, K.A., Venkateswaran, G., 2017. Biological detoxification of Aflatoxin B1 by Bacillus licheniformis CFR1. Food Control 71, 234-241.
Rieswijk, L., Claessen, S.M., Bekers, O., van Herwijnen, M., Theunissen, D.H., Jennen, D.G., de Kok, T.M., Kleinjans, J.C., van Breda, S.G., 2016. Aflatoxin B1 induces persistent epigenomic effects in primary human hepatocytes associated with hepatocellular carcinoma. Toxicology 350, 31-39.
Rogowska, A., Pomastowski, P., Walczak, J., Railean-Plugaru, V., Rudnicka, J., Buszewski, B., 2019. Investigation of zearalenone adsorption and biotransformation by microorganisms cultured under cellular stress conditions. Toxins 11, 463.
Rotimi, O.A., Rotimi, S.O., Duru, C.U., Ebebeinwe, O.J., Abiodun, A.O., Oyeniyi, B.O., Faduyile, F.A., 2017. Acute aflatoxin B1–Induced hepatotoxicity alters gene expression and disrupts lipid and lipoprotein metabolism in rats. Toxicology reports 4, 408-414.
Sadiq, F.A., Yan, B., Tian, F., Zhao, J., Zhang, H., Chen, W., 2019. Lactic acid bacteria as antifungal and anti‐mycotoxigenic agents: a comprehensive review. Comprehensive Reviews in Food Science and Food Safety 18, 1403-1436.
Sandlin, N., Russell Kish, D., Kim, J., Zaccaria, M., Momeni, B., 2022. Current and emerging tools of computational biology to improve the detoxification of mycotoxins. Applied and Environmental Microbiology 88, e02102-02121.
Sangare, L., Zhao, Y., Folly, Y.M.E., Chang, J., Li, J., Selvaraj, J.N., Xing, F., Zhou, L., Wang, Y., Liu, Y., 2014. Aflatoxin B1 degradation by a Pseudomonas strain. Toxins 6, 3028-3040.
Sangsila, A., Faucet-Marquis, V., Pfohl-Leszkowicz, A., Itsaranuwat, P., 2016. Detoxification of zearalenone by Lactobacillus pentosus strains. Food Control 62, 187-192.
Scarpari, M., Bello, C., Pietricola, C., Zaccaria, M., Bertocchi, L., Angelucci, A., Ricciardi, M.R., Scala, V., Parroni, A., Fabbri, A.A., 2014. Aflatoxin control in maize by Trametes versicolor. Toxins 6, 3426-3437.
Schwartz, P., Bucheli, T.D., Wettstein, F.E., Burkhardt‐Holm, P., 2013. Life‐cycle exposure to the estrogenic mycotoxin zearalenone affects zebrafish (Danio rerio) development and reproduction. Environmental Toxicology 28, 276-289.
Shang, L., Bai, X., Chen, C., Liu, L., Li, M., Xia, X., Wang, Y., 2019. Isolation and identification of a Bacillus megaterium strain with ochratoxin A removal ability and antifungal activity. Food Control 106, 106743.
Shang, Q., Yang, Z., Yang, W., Li, Z., Zhang, G., Jiang, S., 2016. Toxicity of mycotoxins from contaminated corn with or withoutyeast cell wall adsorbent on broiler chickens. Asian-Australasian Journal of Animal Sciences 29, 674.
Shetty, P.H., Jespersen, L., 2006. Saccharomyces cerevisiae and lactic acid bacteria as potential mycotoxin decontaminating agents. Trends in food science & technology 17, 48-55.
Shi, L., Liang, Z., Li, J., Hao, J., Xu, Y., Huang, K., Tian, J., He, X., Xu, W., 2014. Ochratoxin A biocontrol and biodegradation by Bacillus subtilis CW 14. Journal of the Science of Food and Agriculture 94, 1879-1885.
Shi, L., Liang, Z., Xu, S., Zheng, H., Huang, K., 2013. Adsorption and degradation of ochratoxin A by Bacillus licheniformis Sl-1. Journal of Agricultural Biotechnology 21, 1420-1425.
Shu, X., Wang, Y., Zhou, Q., Li, M., Hu, H., Ma, Y., Chen, X., Ni, J., Zhao, W., Huang, S., 2018. Biological degradation of aflatoxin B1 by cell-free extracts of Bacillus velezensis DY3108 with broad pH stability and excellent thermostability. Toxins 10, 330.
Shukla, S., Park, J.H., Kim, M., 2020. Efficient, safe, renewable, and industrially feasible strategy employing Bacillus subtilis with alginate bead composite for the reduction of ochratoxin A from wine. Journal of Cleaner Production 242, 118344.
Smith, M.-C., Madec, S., Coton, E., Hymery, N., 2016. Natural co-occurrence of mycotoxins in foods and feeds and their in vitro combined toxicological effects. Toxins 8, 94.
Song, J., Zhang, S., Xie, Y., Li, Q., 2019. Purification and characteristics of an aflatoxin B1 degradation enzyme isolated from Pseudomonas aeruginosa. FEMS microbiology letters 366, fnz034.
Song, Y., Wang, Y., Guo, Y., Qiao, Y., Ma, Q., Ji, C., Zhao, L., 2021. Degradation of zearalenone and aflatoxin B1 by Lac2 from Pleurotus pulmonarius in the presence of mediators. Toxicon 201, 1-8.
Streit, E., Schatzmayr, G., Tassis, P., Tzika, E., Marin, D., Taranu, I., Tabuc, C., Nicolau, A., Aprodu, I., Puel, O., 2012. Current situation of mycotoxin contamination and co-occurrence in animal feed—Focus on Europe. Toxins 4, 788-809.
Taheur, F.B., Fedhila, K., Chaieb, K., Kouidhi, B., Bakhrouf, A., Abrunhosa, L., 2017. Adsorption of aflatoxin B1, zearalenone and ochratoxin A by microorganisms isolated from Kefir grains. International Journal of Food Microbiology 251, 1-7.
Takahashi-Ando, N., Kimura, M., Kakeya, H., Osada, H., Yamaguchi, I., 2002. A novel lactonohydrolase responsible for the detoxification of zearalenone: enzyme purification and gene cloning. Biochemical journal 365, 1-6.
Taroub, B., Salma, L., Manel, Z., Ouzari, H.-I., Hamdi, Z., Moktar, H., 2019. Isolation of lactic acid bacteria from grape fruit: antifungal activities, probiotic properties, and in vitro detoxification of ochratoxin A. Annals of Microbiology 69, 17-27.
Taylor, M.C., Jackson, C.J., Tattersall, D.B., French, N., Peat, T.S., Newman, J., Briggs, L.J., Lapalikar, G.V., Campbell, P.M., Scott, C., 2010. Identification and characterization of two families of F420H2 dependent reductases from Mycobacteria that catalyse aflatoxin degradation. Molecular microbiology 78, 561-575.
Tola, M., Kebede, B., 2016. Occurrence, importance and control of mycotoxins: A review. Cogent Food & Agriculture 2, 1191103. Utermark, J., Karlovsky, P., 2007. Role of zearalenone lactonase in protection of Gliocladium roseum from fungitoxic effects of the mycotoxin zearalenone. Applied and environmental microbiology 73, 637-642.
Varga, J., Frisvad, J.C., Samson, R., 2011. Two new aflatoxin producing species, and an overview of Aspergillus section Flavi. Studies in mycology 69, 57-80.
Vega, M.F., Dieguez, S.N., Riccio, B., Aranguren, S., Giordano, A., Denzoin, L., Soraci, A.L., Tapia, M.O., Ross, R., Apás, A., 2017. Zearalenone adsorption capacity of lactic acid bacteria isolated from pigs. brazilian journal of microbiology 48, 715-723.
Vila-Donat, P., Marín, S., Sanchis, V., Ramos, A., 2018. A review of the mycotoxin adsorbing agents, with an emphasis on their multi-binding capacity, for animal feed decontamination. Food and chemical toxicology 114, 246-259.
Wall-Martínez, H.A., Pascari, X., Bigordà, A., Ramos, A.J., Marín, S., Sanchis, V., 2019. The fate of Fusarium mycotoxins (deoxynivalenol and zearalenone) through wort fermenting by Saccharomyces yeasts (S. cerevisiae and S. pastorianus). Food Research International 126, 108587.
Wang, J., Ogata, M., Hirai, H., Kawagishi, H., 2011. Detoxification of aflatoxin B1 by manganese peroxidase from the white-rot fungus Phanerochaete sordida YK-624. FEMS microbiology letters 314, 164-169.
Wang, J., Xie, Y., 2020. Review on microbial degradation of zearalenone and aflatoxins. Grain & Oil Science and Technology 3, 117-125.
Wang, L., Wu, J., Liu, Z., Shi, Y., Liu, J., Xu, X., Hao, S., Mu, P., Deng, F., Deng, Y., 2019a. Aflatoxin B1 degradation and detoxification by Escherichia coli CG1061 isolated from chicken cecum. Frontiers in pharmacology 9, 1548.
Wang, N., Wu, W., Pan, J., Long, M., 2019b. Detoxification strategies for zearalenone using microorganisms: A review. Microorganisms 7, 208.
Wang, X., Bai, Y., Huang, H., Tu, T., Wang, Y., Wang, Y., Luo, H., Yao, B., Su, X., 2019c. Degradation of aflatoxin B1 and zearalenone by bacterial and fungal laccases in presence of structurally defined chemicals and complex natural mediators. Toxins 11, 609.
Wang, X., Qin, X., Hao, Z., Luo, H., Yao, B., Su, X., 2019d. Degradation of four major mycotoxins by eight manganese peroxidases in presence of a dicarboxylic acid. Toxins 11, 566.
Woźny, M., Dobosz, S., Hliwa, P., Gomułka, P., Król, J., Obremski, K., Blahova, J., Svobodova, Z., Michalik, O., Ocalewicz, K., 2020. Feed-borne exposure to zearalenone impairs reproduction of rainbow trout. Aquaculture 528, 735522.
Wu, N., Ou, W., Zhang, Z., Wang, Y., Xu, Q., Huang, H., 2021. Recent advances in detoxification strategies for zearalenone contamination in food and feed. Chinese Journal of Chemical Engineering 30, 168-177.
Wu, Y.-Z., Lu, F.-P., Jiang, H.-L., Tan, C.-P., Yao, D.-S., Xie, C.-F., Liu, D.-L., 2015. The furofuran-ring selectivity, hydrogen peroxide-production and low Km value are the three elements for highly effective detoxification of aflatoxin oxidase. Food and Chemical Toxicology 76, 125-131.
Xiong, D., Wen, J., Lu, G., Li, T., Long, M., 2022. Isolation, purification, and characterization of a laccase degrading aflatoxin b1 from Bacillus amyloliquefaciens B10. Toxins 14, 250.
Xiong, K., Wang, X.l., Zhi, H.W., Sun, B.G., Li, X.T., 2017. Identification and safety evaluation of a product from the biodegradation of ochratoxin A by an Aspergillus strain. Journal of the Science of Food and Agriculture 97, 434-443.
Xiong, K., Zhi, H.-w., Liu, J.-y., Wang, X.-y., Zhao, Z.-y., Pei, P.-g., Deng, L., Xiong, S.-y., 2021. Detoxification of Ochratoxin A by a novel Aspergillus oryzae strain and optimization of its biodegradation. Revista argentina de microbiología 53, 48-58.
Xu, H., Wang, L., Sun, J., Wang, L., Guo, H., Ye, Y., Sun, X., 2022. Microbial detoxification of mycotoxins in food and feed. Critical reviews in food science and nutrition 62, 4951-4969.
Xu, J., Wang, H., Zhu, Z., Ji, F., Yin, X., Hong, Q., Shi, J., 2016. Isolation and characterization of Bacillus amyloliquefaciens ZDS-1: Exploring the degradation of Zearalenone by Bacillus spp. Food Control 68, 244-250.
Xu, X., Pang, M., Liu, J., Wang, Y., Wu, X., Huang, K., Liang, Z., 2021. Genome mining reveals the genes of carboxypeptidase for OTA-detoxification in Bacillus subtilis CW14. International journal of biological macromolecules 186, 800-810.
Yi, P.-J., Pai, C.-K., Liu, J.-R., 2011. Isolation and characterization of a Bacillus licheniformis strain capable of degrading zearalenone. World Journal of Microbiology and Biotechnology 27, 1035-1043.
Yiannikouris, A., FRANCois, J., Poughon, L., Dussap, C.-G., Bertin, G., Jeminet, G., Jouany, J.-P., 2004. Alkali extraction of β-D-glucans from Saccharomyces cerevisiae cell wall and study of their adsorptive properties toward zearalenone. Journal of Agricultural and Food Chemistry 52, 3666-3673.
Yu, Y., Qiu, L., Wu, H., Tang, Y., Lai, F., Yu, Y., 2011a. Oxidation of zearalenone by extracellular enzymes from Acinetobacter sp. SM04 into smaller estrogenic products. World Journal of Microbiology and Biotechnology 27, 2675-2681.
Yu, Y., Qiu, L., Wu, H., Tang, Y., Yu, Y., Li, X., Liu, D., 2011b. Degradation of zearalenone by the extracellular extracts of Acinetobacter sp. SM04 liquid cultures. Biodegradation 22, 613-622.
Zhang, H., Dong, M., Yang, Q., Apaliya, M.T., Li, J., Zhang, X., 2016. Biodegradation of zearalenone by Saccharomyces cerevisiae: Possible involvement of ZEN responsive proteins of the yeast. Journal of Proteomics 143, 416-423.
Zhang, Y., Li, Z., Lu, Y., Zhang, J., Sun, Y., Zhou, J., Tu, T., Gong, W., Sun, W., Wang, Y., 2022. Characterization of Bacillus velezensis E2 with abilities to degrade ochratoxin A and biocontrol against Aspergillus westerdijkiae fc-1. Toxicon 216, 125-131.
Zhang, Y., Wu, D., Su, Y., Xie, B., 2021. Occurrence, influence and removal strategies of mycotoxins, antibiotics and microplastics in anaerobic digestion treating food waste and co-digestive biosolids: a critical review. Bioresource Technology 330, 124987.
Zhao, M., Wang, X., Xu, S., Yuan, G., Shi, X., Liang, Z., 2020. Degradation of ochratoxin A by supernatant and ochratoxinase of Aspergillus niger W-35 isolated from cereals. World Mycotoxin Journal 13, 287-298.




 Prevención de micotoxicosis
Prevención de micotoxicosis