ANTECEDENTES
ANTECEDENTES
Muchas especies fúngicas que crecen en los cereales y piensos producen metabolitos secundarios denominados micotoxinas.
Las micotoxinas, como la fumonisina FB1 (FB1) y la toxina T-2 (Figura 1), son el mayor reto para los productores de piensos y, por tanto, es necesario realizar un seguimiento y remediación regular, ya que estas toxinas pueden ser muy perjudiciales para las personas y los animales.
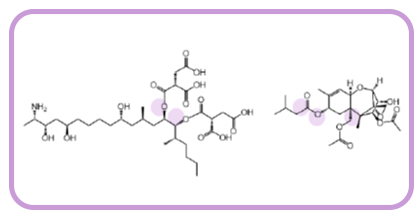
Figura 1. FB1 y toxina T-2. Los sitios que pueden ser potencialmente atacados por enzimas están resaltados en lila.
 Se calcula que las pérdidas económicas atribuidas a las micotoxinas en el sector agroalimentario ascienden a miles de millones de dólares, por lo que el potencial económico de las medidas de protección o eliminación de micotoxinas es igualmente elevado.
Se calcula que las pérdidas económicas atribuidas a las micotoxinas en el sector agroalimentario ascienden a miles de millones de dólares, por lo que el potencial económico de las medidas de protección o eliminación de micotoxinas es igualmente elevado.
Hoy en día, ya existen diferentes medidas disponibles, como los enfoques mecánicos, térmicos o químicos.
 Sin embargo, estos enfoques tienen como desventaja asociada la posibilidad de generar productos secundarios desconocidos o no deseados que pueden resultar tan peligrosos como la toxina original.
Sin embargo, estos enfoques tienen como desventaja asociada la posibilidad de generar productos secundarios desconocidos o no deseados que pueden resultar tan peligrosos como la toxina original.
Las enzimas son proteínas que catalizan reacciones bioquímicas que hacen posible la vida desde hace 3.800 millones de años.
 Los científicos han sido capaces de “domesticarlas” de tal manera que las enzimas son ahora actores clave para la economía circular y para el procesamiento, fabricación y consumo de productos cotidianos, tanto para humanos como para animales2.
Los científicos han sido capaces de “domesticarlas” de tal manera que las enzimas son ahora actores clave para la economía circular y para el procesamiento, fabricación y consumo de productos cotidianos, tanto para humanos como para animales2.

OBJETIVO
OBJETIVO
Dotar a la industria agroalimentaria de enzimas capaces de eliminar eficazmente las micotoxinas puede contribuir a mantener la seguridad alimentaria y de los piensos, así como reducir la transferencia de micotoxinas de los animales a los seres humanos.
Sin embargo, la opción de encontrar esas nuevas enzimas, en particular las que degradan micotoxinas como FB1 y toxina T-2, es difícil, costosa y requiere mucho tiempo.
 En realidad, solo se conocen unas pocas enzimas que degraden ambas toxinas1.
En realidad, solo se conocen unas pocas enzimas que degraden ambas toxinas1.
 En esta contribución, ofrecemos una visión global de nuestra plataforma sinérgica para mejorar la bioprospección de metagenomas medioambientales en busca de estas nuevas enzimas raras.
En esta contribución, ofrecemos una visión global de nuestra plataforma sinérgica para mejorar la bioprospección de metagenomas medioambientales en busca de estas nuevas enzimas raras.
Además, aportamos pruebas experimentales que demuestran la degradación eficaz de FB1 y toxina T-2 mediante dos nuevas hidrolasas recuperadas a través de cribados naïve y bioinformáticos.
MATERIALES & MÉTODOS
MATERIALES & MÉTODOS
La opción de encontrar nuevas enzimas, aunque difícil, costosa (30.000 euros/enzima) y lenta (15 meses/enzima), es realista gracias a los recientes avances tecnológicos.
Para aprovechar plenamente el potencial de los datos de secuenciación y como fuente de nuevas enzimas es esencial combinar:
- ⇰ Bioinformática
- ⇰ Machine-learning
- ⇰ Predicción precisa de la estructura de las proteínas
- ⇰ Técnicas de inteligencia artificial basadas en datos
- ⇰ Cribados naïve con sustratos apropiados
Con el fin de acceder a nuevas enzimas capaces de degradar micotoxinas, hemos establecido una plataforma global y sinérgica para mejorar la bioprospección de metagenomas medioambientales en busca de nuevas enzimas raras (Figura 2).
Consiste en los siguientes pasos:
- 1. Extracción y secuenciación del ADN de las comunidades microbianas (el llamado metagenoma) de un gran número de muestras ambientales.
- 2. Procesamiento bioinformático de las secuencias representativas de los metagenomas para filtrar aquellas enzimas del metagenoma con potencial para degradar FB1 y toxina T-2, concretamente, hidrolasas.
- 3. Análisis computacional de acoplamiento molecular para proporcionar además análisis computacional de mecánica cuántica/ molecular (QM/MM) para identificar, mediante la cuantificación de los eventos catalíticos, cuáles de las enzimas seleccionadas pueden unirse y potencialmente degradar FB1 y toxina T-2.
- 4. Por último, la validación experimental: síntesis genética, producción, purificación y caracterización.
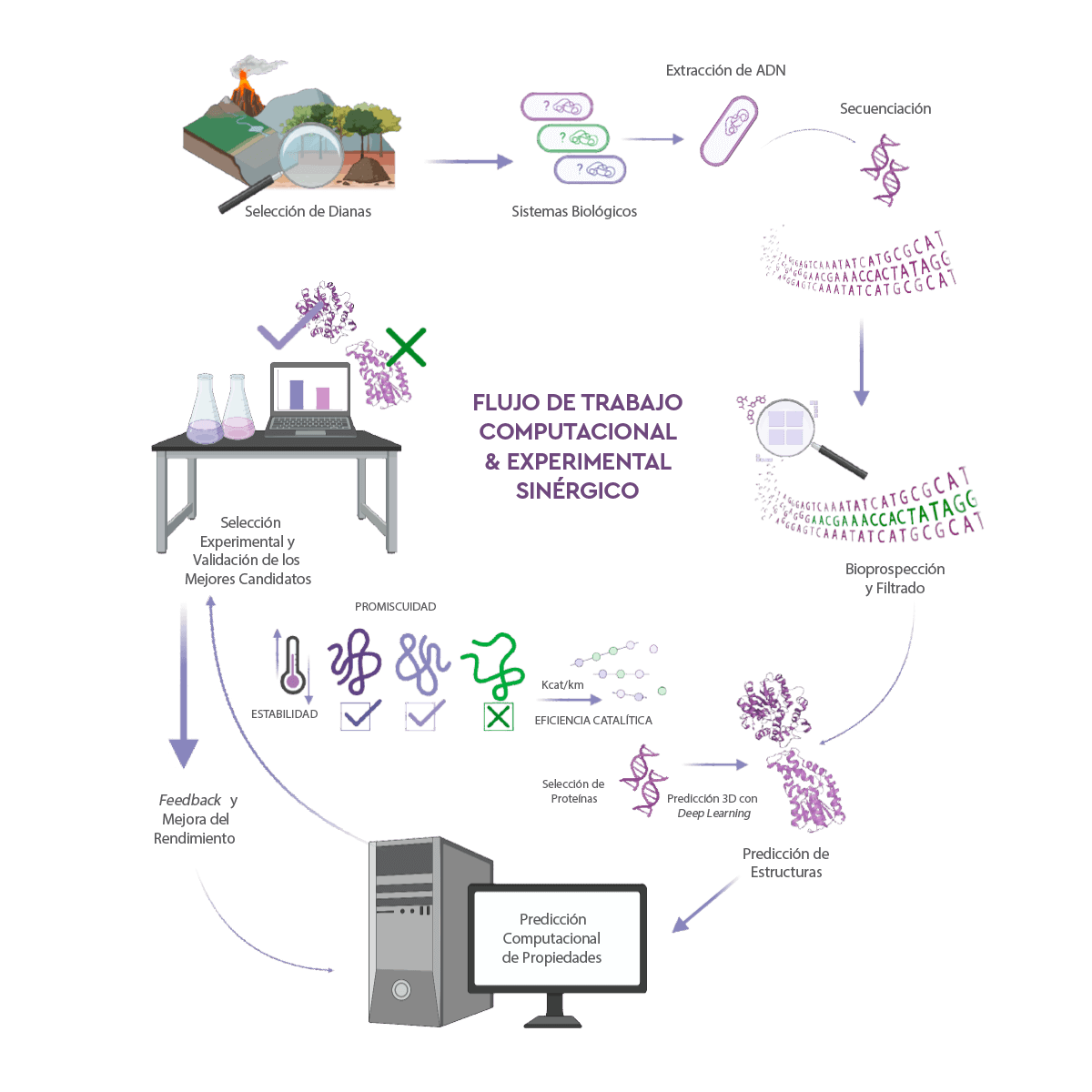
Figura 2. Flujo de trabajo esquemático para la bioprospección de enzimas capaces de degradar toxinas. Se muestran los pasos relacionados con la extracción, secuenciación, ensamblaje, anotación y cribado virtual de nuevas enzimas a partir de metagenomas de muestras ambientales, seguidos de su predicción precisa de la estructura proteica, caracterización de alto rendimiento y mejora iterativa mediante ingeniería (creado con BioRender.com).
RESULTADOS
RESULTADOS
Mediante la aplicación de nuestra plataforma sinérgica, hemos bioprospeccionado con éxito metagenomas medioambientales y hemos encontrado varias hidrolasas degradadoras de FB1 y toxina T-2, a destacar:
- ✓ Una esterasa de la familia VIII, aislada a partir del ADN metagenómico de comunidades microbianas que habitan la zona costera del puerto de Milazzo (Sicilia, Italia), capaz de degradar la toxina T-2 (Figura 3) sin producir la toxina intermediaria HT-21.
- ✓ Una esterasa capaz de degradar 2-10 ppm de FB1 (100% de degradación) tras un tiempo de reacción de 5 minutos a concentraciones tan bajas como 5 μg/ml a 37-90 °C3.
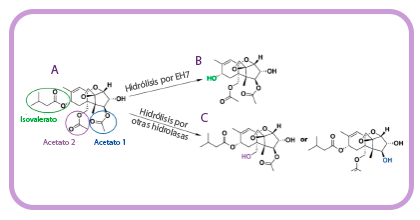
Figura 3. (A) Estructura de la toxina T-2. Los diferentes enlaces éster que pueden hidrolizarse están coloreados. Producto(s) obtenido(s) por la degradación de la toxina T-2 a través del grupo isovalerato por la esterasa EH7 (B) y por la hidrólisis de ambos acetatos por diferentes hidrolasas (C).
Autores
Manuel Ferrer1, David Almendral1, Jog Raj2, Hunor Farkaš2 y Marko Vasiljević2
1Departamento de Biocatálisis Aplicada, ICP, CSIC, Marie Curie 2, Madrid, España
2PATENT CO, DOO., Vlade Ćetković 1A, 24 211 Mišićevo, Serbia
1. Cea-Rama I, Coscolín C, Gonzalez-Alfonso JL, Raj J, Vasiljević M, Plou FJ, Ferrer M, Sanz-Aparicio J. FEBS J. 2022;289(21):6714-6730.
2. Molina-Espeja, P. et al. Oxford Open Climate Change, 2023; kgad003, https://doi.org/10.1093/oxfclm/kgad003
3. Fumonisin removal by enzymatic detoxification – EP1641.1745




 Prevención de micotoxicosis
Prevención de micotoxicosis